„Aminosäuren“ – Versionsunterschied
| [ungesichtete Version] | [gesichtete Version] |
K Änderungen von 2003:4F:4C20:6400:E045:9B4D:6DFB:F61B (Diskussion) auf die letzte Version von O0TsRVi7 zurückgesetzt |
|||
| Zeile 11: | Zeile 11: | ||
== Geschichte == |
== Geschichte == |
||
[[Datei:Aminosaeuren.png|mini|hochkant=2.4|[[Strukturformel]]n von 20 natürlichen α-Aminosäuren]] |
|||
Die erste Aminosäure wurde 1805 im Pariser Labor von [[Louis-Nicolas Vauquelin]] und dessen Schüler [[Pierre Jean Robiquet]] aus dem Saft von [[Gemüsespargel|Spargel]] (''Asparagus officinalis'') isoliert und danach [[Asparagin]] genannt.<ref>L. Vauquelin, P. Robiquet: ''The discovery of a new plant principle in Asparagus sativus.'' In: ''Annales de Chimie.'' Band 57, 1806, S. 88–93.</ref> 1931 wurde als letzte der üblichen proteinaufbauenden Aminosäuren das [[Threonin]] im [[Fibrin]] entdeckt und 1935 seiner Struktur nach geklärt von [[William Rose (Chemiker)|William Rose]]. Rose hatte durch Experimente mit verschiedenen Futtermitteln herausgefunden, dass die bis dato entdeckten 19 Aminosäuren als Zusatz nicht ausreichten.<ref>W. Rose et al.: ''Feeding Experiments with Mixtures of Highly Purified Amino Acids. VIII. Isolation and Identification of a New Essential Amino Acid.'' In: ''Journal of Biological Chemistry.'' Band 112, 1935, S. 283–302.</ref> Er stellte auch die [[Essentieller Stoff|Essentialität]] anderer Aminosäuren fest und ermittelte je die für ein optimales Wachstum mindestens [[Recommended Daily Allowance|erforderliche Tagesdosis]].<ref>R. Simoni, R. Hill, M. Vaughan: [http://www.jbc.org/content/277/37/e25 ''The Discovery of the Amino Acid Threonine: the Work of William C. Rose.''] In: ''Journal of Biological Chemistry.'' Band 277, Nr. 37, 13 September 2002, S. 56–58.</ref> |
Die erste Aminosäure wurde 1805 im Pariser Labor von [[Louis-Nicolas Vauquelin]] und dessen Schüler [[Pierre Jean Robiquet]] aus dem Saft von [[Gemüsespargel|Spargel]] (''Asparagus officinalis'') isoliert und danach [[Asparagin]] genannt.<ref>L. Vauquelin, P. Robiquet: ''The discovery of a new plant principle in Asparagus sativus.'' In: ''Annales de Chimie.'' Band 57, 1806, S. 88–93.</ref> 1931 wurde als letzte der üblichen proteinaufbauenden Aminosäuren das [[Threonin]] im [[Fibrin]] entdeckt und 1935 seiner Struktur nach geklärt von [[William Rose (Chemiker)|William Rose]]. Rose hatte durch Experimente mit verschiedenen Futtermitteln herausgefunden, dass die bis dato entdeckten 19 Aminosäuren als Zusatz nicht ausreichten.<ref>W. Rose et al.: ''Feeding Experiments with Mixtures of Highly Purified Amino Acids. VIII. Isolation and Identification of a New Essential Amino Acid.'' In: ''Journal of Biological Chemistry.'' Band 112, 1935, S. 283–302.</ref> Er stellte auch die [[Essentieller Stoff|Essentialität]] anderer Aminosäuren fest und ermittelte je die für ein optimales Wachstum mindestens [[Recommended Daily Allowance|erforderliche Tagesdosis]].<ref>R. Simoni, R. Hill, M. Vaughan: [http://www.jbc.org/content/277/37/e25 ''The Discovery of the Amino Acid Threonine: the Work of William C. Rose.''] In: ''Journal of Biological Chemistry.'' Band 277, Nr. 37, 13 September 2002, S. 56–58.</ref> |
||
In der Zeit zwischen 1805 und 1935 waren viele der damals bekannten Chemiker und Pharmazeuten daran beteiligt, Aminosäuren erstmalig zu isolieren sowie deren Struktur aufzuklären. So gelang [[Emil Fischer]], auf den auch die [[Fischer-Projektion]] zurückgeht, die finale Aufklärung der Struktur von [[Serin]] (1901), [[Lysin]] (1902), [[Valin]] (1906) und [[Cystein]] (1908). Auch [[Albrecht Kossel]] (1896 [[Histidin]] aus Störsperma), [[Richard Willstätter]] (1900 [[Prolin]] via Synthese) und [[Frederick Hopkins]] (1901 [[Tryptophan]] aus [[Casein]]) wurden später Nobelpreisträger. Der deutsche Chemiker [[Ernst Schulze (Chemiker)|Ernst Schulze]] isolierte drei Aminosäuren erstmals – 1877 [[Glutamin]] aus Rüben, 1881 [[Phenylalanin]] und 1886 [[Arginin]] aus [[Lupinen]]– und war an der Strukturaufklärung weiterer Aminosäuren beteiligt. Zuvor hatte [[Heinrich Ritthausen]] 1866 [[Glutaminsäure]] aus Getreideeiweiß, dem [[Gluten]], kristallin gewonnen. [[Wilhelm Dittmar]] klärte 1872 die Struktur von Glutamin und Glutaminsäure, deren Salze [[Glutamate]] sind. |
In der Zeit zwischen 1805 und 1935 waren viele der damals bekannten Chemiker und Pharmazeuten daran beteiligt, Aminosäuren erstmalig zu isolieren sowie deren Struktur aufzuklären. So gelang [[Emil Fischer]], auf den auch die [[Fischer-Projektion]] zurückgeht, die finale Aufklärung der Struktur von [[Serin]] (1901), [[Lysin]] (1902), [[Valin]] (1906) und [[Cystein]] (1908). Auch [[Albrecht Kossel]] (1896 [[Histidin]] aus Störsperma), [[Richard Willstätter]] (1900 [[Prolin]] via Synthese) und [[Frederick Hopkins]] (1901 [[Tryptophan]] aus [[Casein]]) wurden später Nobelpreisträger. Der deutsche Chemiker [[Ernst Schulze (Chemiker)|Ernst Schulze]] isolierte drei Aminosäuren erstmals – 1877 [[Glutamin]] aus Rüben, 1881 [[Phenylalanin]] und 1886 [[Arginin]] aus [[Lupinen]]– und war an der Strukturaufklärung weiterer Aminosäuren beteiligt. Zuvor hatte [[Heinrich Ritthausen]] 1866 [[Glutaminsäure]] aus Getreideeiweiß, dem [[Gluten]], kristallin gewonnen. [[Wilhelm Dittmar]] klärte 1872 die Struktur von Glutamin und Glutaminsäure, deren Salze [[Glutamate]] sind. |
||
[[Datei:Aminosaeuren.png|mini|hochkant=2.0|[[Strukturformel]]n von zwanzig natürlichen α-Aminosäuren]] |
|||
Bereits 1819 hatte [[Henri Braconnot]] das [[Glycin]] aus Leim und [[Joseph Louis Proust]] das [[Leucin]] aus Getreide isoliert. 1820 entdeckte [[William Hyde Wollaston]] das [[schwefel]]haltige [[Cystin]] in [[Blasenstein]]en, doch erst 1884 [[Eugen Baumann]] das monomere [[Cystein]]. [[Eugen von Gorup-Besánez]] isolierte 1856 das [[Valin]] aus [[Bauchspeicheldrüse|Pankreassaft]]. 1846 hatte [[Justus von Liebig]] aus Casein erstmals das [[Tyrosin]] abtrennen können, dessen Struktur 1869 [[Ludwig Barth zu Barthenau|Ludwig von Barth]] klärte. Im [[Hydrolysat]] des Casein entdeckte [[Edmund Drechsel]] 1889 auch das [[Lysin]] und später [[John Howard Mueller]] 1922 das schwefelhaltige [[Methionin]] als 19. Aminosäure, deren [[Strukturformel]] [[George Barger]] und Philip Coine 1928 angaben. In [[Melasse]] hatte [[Felix Ehrlich (Biochemiker)|Felix Ehrlich]] schon 1903 als 18. das [[Isoleucin]] gefunden, ein [[Isomerie#Konstitutionsisomerie oder Strukturisomerie|Strukturisomer]] des Leucin. |
Bereits 1819 hatte [[Henri Braconnot]] das [[Glycin]] aus Leim und [[Joseph Louis Proust]] das [[Leucin]] aus Getreide isoliert. 1820 entdeckte [[William Hyde Wollaston]] das [[schwefel]]haltige [[Cystin]] in [[Blasenstein]]en, doch erst 1884 [[Eugen Baumann]] das monomere [[Cystein]]. [[Eugen von Gorup-Besánez]] isolierte 1856 das [[Valin]] aus [[Bauchspeicheldrüse|Pankreassaft]]. 1846 hatte [[Justus von Liebig]] aus Casein erstmals das [[Tyrosin]] abtrennen können, dessen Struktur 1869 [[Ludwig Barth zu Barthenau|Ludwig von Barth]] klärte. Im [[Hydrolysat]] des Casein entdeckte [[Edmund Drechsel]] 1889 auch das [[Lysin]] und später [[John Howard Mueller]] 1922 das schwefelhaltige [[Methionin]] als 19. Aminosäure, deren [[Strukturformel]] [[George Barger]] und Philip Coine 1928 angaben. In [[Melasse]] hatte [[Felix Ehrlich (Biochemiker)|Felix Ehrlich]] schon 1903 als 18. das [[Isoleucin]] gefunden, ein [[Isomerie#Konstitutionsisomerie oder Strukturisomerie|Strukturisomer]] des Leucin. |
||
| Zeile 44: | Zeile 44: | ||
== Aufbau der Aminosäuren == |
== Aufbau der Aminosäuren == |
||
{| class="wikitable float- |
{| class="wikitable float-right" width="5%" style="text-align:center" |
||
|- |
|- |
||
| [[Datei:Carbamide Acid Formula V2.svg|center|90px|Carbamidsäure]] <small> Carbamidsäure </small> |
| [[Datei:Carbamide Acid Formula V2.svg|center|90px|Carbamidsäure]] <small> Carbamidsäure </small> |
||
| Zeile 50: | Zeile 50: | ||
|} |
|} |
||
Aminosäuren bestehen aus mindestens zwei Kohlenstoffatomen. Die instabile [[Carbamidsäure]] besitzt lediglich ein Kohlenstoffatom und ist damit keine Aminosäure, sondern ein Kohlensäureamid. Aminosäuren lassen sich in Klassen einteilen je nach dem Kohlenstoffatom, an dem sich die [[Aminogruppe]] relativ zur Carboxygruppe befindet. Sind im [[Molekül]] mehrere Aminogruppen vertreten, so bestimmt das Kohlenstoffatom, dessen Aminogruppe dem Carboxy-Kohlenstoff am nächsten steht, um welche Klasse von Aminosäuren es sich handelt. |
|||
{| class="wikitable float-right" style="text-align:center" |
|||
{| class="wikitable float-right" width="25%" style="text-align:center" |
|||
|- class="hintergrundfarbe6" |
|- class="hintergrundfarbe6" |
||
| Allgemeine Struktur von<br |
| Allgemeine Struktur von Aminosäuren<br />(R = Seitenkette) |
||
|- |
|- |
||
| [[Datei:Alpha-Amino Acid Formula V2.svg|center|115px]] <small>α-Aminosäure</small> |
| [[Datei:Alpha-Amino Acid Formula V2.svg|center|115px]] <small>α-Aminosäure</small> |
||
| Zeile 62: | Zeile 64: | ||
|- |
|- |
||
|} |
|} |
||
Aminosäuren bestehen aus mindestens zwei Kohlenstoffatomen. Die instabile [[Carbamidsäure]] besitzt lediglich ein Kohlenstoffatom und ist damit keine Aminosäure, sondern ein Kohlensäureamid. Aminosäuren lassen sich in Klassen einteilen je nach dem Kohlenstoffatom, an dem sich die [[Aminogruppe]] relativ zur Carboxygruppe befindet. Sind im [[Molekül]] mehrere Aminogruppen vertreten, so bestimmt das Kohlenstoffatom, dessen Aminogruppe dem Carboxy-Kohlenstoff am nächsten steht, um welche Klasse von Aminosäuren es sich handelt. |
|||
* ''α-Aminosäuren:'' Die Aminogruppe der α-Aminosäuren befindet sich am zweiten Kohlenstoffatom, einschließlich des Carboxy-Kohlenstoffatoms. Die Zählung beginnt immer mit dem Carboxy-Kohlenstoff. Die IUPAC-Bezeichnung lautet daher 2-Aminocarbonsäuren. Der einfachste Vertreter der α-Aminosäuren ist die proteinogene Aminosäure [[Glycin]].<br />Der Begriff Aminosäuren wird meist synonym für eine Gruppe von α-Aminosäuren verwendet, die hauptsächlich aus <small>L</small>-α-Aminosäuren besteht: die ''proteinogenen Aminosäuren''. Die proteinogenen Aminosäuren sind die Bausteine sämtlicher Proteine allen Lebens auf der Erde und neben den [[Nukleinsäure]]n ''Grundbausteine des Lebens''. |
* ''α-Aminosäuren:'' Die Aminogruppe der α-Aminosäuren befindet sich am zweiten Kohlenstoffatom, einschließlich des Carboxy-Kohlenstoffatoms. Die Zählung beginnt immer mit dem Carboxy-Kohlenstoff. Die IUPAC-Bezeichnung lautet daher 2-Aminocarbonsäuren. Der einfachste Vertreter der α-Aminosäuren ist die proteinogene Aminosäure [[Glycin]].<br />Der Begriff Aminosäuren wird meist synonym für eine Gruppe von α-Aminosäuren verwendet, die hauptsächlich aus <small>L</small>-α-Aminosäuren besteht: die ''proteinogenen Aminosäuren''. Die proteinogenen Aminosäuren sind die Bausteine sämtlicher Proteine allen Lebens auf der Erde und neben den [[Nukleinsäure]]n ''Grundbausteine des Lebens''. |
||
| Zeile 73: | Zeile 74: | ||
== Säure- und Base-Verhalten == |
== Säure- und Base-Verhalten == |
||
[[Datei:Titration Glycin.png|mini|hochkant=1. |
[[Datei:Titration Glycin.png|mini|hochkant=1.4|Die Abhängigkeit der durchschnittlichen Nettoladung des Glycins vom [[pH-Wert]]]] |
||
Als Feststoffe und in neutralen wässrigen Lösungen liegen Aminosäuren als [[Zwitterion]]en vor, das heißt die Aminogruppe ist protoniert und die Carboxygruppe ist deprotoniert. |
Als Feststoffe und in neutralen wässrigen Lösungen liegen Aminosäuren als [[Zwitterion]]en vor, das heißt die Aminogruppe ist protoniert und die Carboxygruppe ist deprotoniert. |
||
| Zeile 110: | Zeile 111: | ||
|+ Die 20 kanonischen Aminosäuren |
|+ Die 20 kanonischen Aminosäuren |
||
|- class="hintergrundfarbe6" |
|- class="hintergrundfarbe6" |
||
! Aminosäure !! Dreibuchstabencode !! Einbuchstabencode !! Acyl-Gruppe<ref name="IUPAC" /> !! Bemerkung !! Ø in Proteinen<ref name="Bruice">Paula Yurkanis Bruice: ''Organic Chemistry.'' Pearson Education Inc., 2004, 4. Auflage, S. 960–962, ISBN 0-13-121730-5.</ref> |
|||
! rowspan="2" | Amino-<br>säure |
|||
! Drei- |
|||
! Ein- |
|||
! rowspan="2" | Acyl-<br>Gruppe<ref name="IUPAC" /> |
|||
! rowspan="2" | Bemerkung |
|||
! rowspan="2" | Ø in<br>Proteinen<ref name="Bruice">Paula Yurkanis Bruice: ''Organic Chemistry.'' Pearson Education Inc., 2004, 4. Auflage, S. 960–962, ISBN 0-13-121730-5.</ref> |
|||
|- |
|||
! colspan="2" style="background-color:#B3B7FF;" | buchstabencode |
|||
|- |
|- |
||
| [[Alanin]] || Ala || A || Alanyl- || [[Essentielle Aminosäure|nicht-essentiell]] || 9,0 % |
| [[Alanin]] || Ala || A || Alanyl- || [[Essentielle Aminosäure|nicht-essentiell]] || 9,0 % |
||
| Zeile 166: | Zeile 160: | ||
{| class="wikitable" |
{| class="wikitable" |
||
|- |
|||
|- class="hintergrundfarbe6" |
|||
! |
! Platzhalter |
||
! Dreibuchstabencode |
|||
! Drei- |
|||
! Einbuchstabencode |
|||
! Ein- |
|||
|- class="hintergrundfarbe6" |
|||
! colspan="2" class="hintergrundfarbe6" | buchstabencode |
|||
|- style="text-align:center;" |
|- style="text-align:center;" |
||
| Asparagin oder Asparaginsäure |
| Asparagin oder Asparaginsäure |
||
| Zeile 186: | Zeile 178: | ||
|- style="text-align:center;" |
|- style="text-align:center;" |
||
| unbekannte Aminosäure |
| unbekannte Aminosäure |
||
| Xaa |
| Xaa (selten Unk) |
||
| X |
| X |
||
|} |
|} |
||
=== Nichtkanonische Aminosäuren === |
=== Nichtkanonische Aminosäuren === |
||
Zu den natürlich vorkommenden Aminosäuren gehören außer den kanonischen die übrigen als '''nichtkanonische Aminosäuren''', wozu proteinogene und nicht-proteinogene zählen. Hierbei lassen sich mehrere Gruppen unterscheiden: |
|||
[[Datei:L-Selenocysteine.svg|mini|hochkant=0.7|L-Selenocystein]] |
|||
{| class="wikitable float-right" |
|||
[[Datei:Pyrrolysine.svg|mini|hochkant=0.7|L-Pyrrolysin]] |
|||
* Zur ersten Gruppe gehören jene '''proteinogenen''' Aminosäuren, die durch eine ''Recodierung'' des genetischen Materials in Proteine eingebaut werden. Die 21. und die 22. proteinogene Aminosäure – [[Selenocystein]] und [[Pyrrolysin]] – gehören hierzu. Für beide Aminosäuren wurden spezifische [[tRNA]]s – tRNA<sup>Sec</sup> bzw. tRNA<sup>Pyl</sup> – gefunden, mit denen die Aminosäure jeweils für den ko-translationalen Einbau am Ribosom bereitgehalten wird. Deren [[Anticodon]] paart, abhängig von Strukturelementen im Kontext der mRNA, mit dem [[Codon]] <tt>UGA</tt> beziehungsweise <tt>UAG</tt>; im Standardcode stellen diese ein [[Stopcodon]] dar. Doch nicht alle Organismen verwenden die nichtkanonischen proteinogenen Aminosäuren dieser Gruppe. |
|||
{| class="wikitable" |
|||
|- class="hintergrundfarbe6" |
|- class="hintergrundfarbe6" |
||
! Aminosäure |
|||
! rowspan="2" | Amino-<br>säure |
|||
! Dreibuchstabencode |
|||
! Drei- |
|||
! Einbuchstabencode |
|||
! Ein- |
|||
|- class="hintergrundfarbe6" |
|||
! colspan="2" class="hintergrundfarbe6" | buchstabencode |
|||
|- style="text-align:center;" |
|- style="text-align:center;" |
||
| [[Pyrrolysin]] |
| [[Pyrrolysin]] |
||
| Zeile 208: | Zeile 202: | ||
| U |
| U |
||
|} |
|} |
||
[[Datei:(S)-N-Formylmethionine V.1.svg|mini|hochkant=0.7|L-''N''-Formylmethionin]] |
|||
Zu den natürlich vorkommenden Aminosäuren gehören außer den kanonischen die übrigen als '''nichtkanonische Aminosäuren''', wozu proteinogene und nicht-proteinogene zählen. Hierbei lassen sich mehrere Gruppen unterscheiden: |
|||
[[Datei:L-Selenocysteine.svg|mini|160px|L-Selenocystein]] |
|||
[[Datei:Pyrrolysine.svg|mini|330px||L-Pyrrolysin]] |
|||
* Zur ersten Gruppe gehören jene '''proteinogenen''' Aminosäuren, die durch eine ''Recodierung'' des genetischen Materials in Proteine eingebaut werden. Die 21. und die 22. proteinogene Aminosäure – [[Selenocystein]] und [[Pyrrolysin]] – gehören hierzu. Für beide Aminosäuren wurden spezifische [[tRNA]]s – tRNA<sup>Sec</sup> bzw. tRNA<sup>Pyl</sup> – gefunden, mit denen die Aminosäure jeweils für den ko-translationalen Einbau am Ribosom bereitgehalten wird. Deren [[Anticodon]] paart, abhängig von Strukturelementen im Kontext der mRNA, mit dem [[Codon]] <tt>UGA</tt> beziehungsweise <tt>UAG</tt>; im Standardcode stellen diese ein [[Stopcodon]] dar. Doch nicht alle Organismen verwenden die nichtkanonischen proteinogenen Aminosäuren dieser Gruppe. |
|||
[[Datei:(S)-N-Formylmethionine V.1.svg|mini|195px|L-''N''-Formylmethionin]] |
|||
: Das übliche [[Startcodon]] <tt>AUG</tt> codiert für die Aminosäure [[Methionin]]. [[Bakterien]] verfügen neben der tRNA<sup>Met</sup> über eine besondere tRNA<sup>fMet</sup>, die ebenfalls mit Methionin beladen wird und als [[tRNA#Initiator tRNAs|Initiator-tRNA]] dient. Die an tRNA<sub>i</sub><sup>fMet</sup> gebundene Aminosäure aber wird am [[N-Terminus|''N''-Terminus]] [[Formylierung|formyliert]] zu ''N''-Formylmethionin (fMet), noch bevor sie bei der [[Translation (Biologie)#Initiation der Translation bei Prokaryoten|Initation]] am Ribosom zur ersten Aminosäure einer Peptidkette werden kann. Dieses Aminosäurederivat Formylmethionin wird daher gelegentlich auch als (23.) proteinogene Aminosäure gezählt. Auch [[Mitochondriales Ribosom#Mitochondriale Translation|Mitochondrien]] und [[Chloroplast#Herkunft der Chloroplasten – Endosymbiontentheorie|Chloroplasten]] nutzen fMet initial. Dagegen wird es im [[Cytosol]] [[Eukaryota|eukaryotischer]] Zellen und in [[Archaeen]] nicht bei der Translation verwendet.<ref name="translation">Katsura Asano: ''Why is start codon selection so precise in eukaryotes?.'' In: ''Translation.'' Band 2, Nr. 1, März 2014, {{DOI|10.4161/trla.28387}}, {{PMC|4705826}}.</ref> |
: Das übliche [[Startcodon]] <tt>AUG</tt> codiert für die Aminosäure [[Methionin]]. [[Bakterien]] verfügen neben der tRNA<sup>Met</sup> über eine besondere tRNA<sup>fMet</sup>, die ebenfalls mit Methionin beladen wird und als [[tRNA#Initiator tRNAs|Initiator-tRNA]] dient. Die an tRNA<sub>i</sub><sup>fMet</sup> gebundene Aminosäure aber wird am [[N-Terminus|''N''-Terminus]] [[Formylierung|formyliert]] zu ''N''-Formylmethionin (fMet), noch bevor sie bei der [[Translation (Biologie)#Initiation der Translation bei Prokaryoten|Initation]] am Ribosom zur ersten Aminosäure einer Peptidkette werden kann. Dieses Aminosäurederivat Formylmethionin wird daher gelegentlich auch als (23.) proteinogene Aminosäure gezählt. Auch [[Mitochondriales Ribosom#Mitochondriale Translation|Mitochondrien]] und [[Chloroplast#Herkunft der Chloroplasten – Endosymbiontentheorie|Chloroplasten]] nutzen fMet initial. Dagegen wird es im [[Cytosol]] [[Eukaryota|eukaryotischer]] Zellen und in [[Archaeen]] nicht bei der Translation verwendet.<ref name="translation">Katsura Asano: ''Why is start codon selection so precise in eukaryotes?.'' In: ''Translation.'' Band 2, Nr. 1, März 2014, {{DOI|10.4161/trla.28387}}, {{PMC|4705826}}.</ref> |
||
| Zeile 228: | Zeile 215: | ||
=== Biochemische Bedeutung === |
=== Biochemische Bedeutung === |
||
==== Aminosäuren als Bausteine von Proteinen ==== |
==== Aminosäuren als Bausteine von Proteinen ==== |
||
[[Datei:Overview proteinogenic amino acids-DE.svg|mini|hochkant=2. |
[[Datei:Overview proteinogenic amino acids-DE.svg|mini|hochkant=2.0|Die natürlich vorkommenden 20 proteinogenen Standard-Aminosäuren, gruppiert nach physikalisch-chemischen Eigenschaften]] |
||
<small>L</small>-Aminosäuren sind in der [[Biochemie]] von großer Bedeutung, da sie die Bausteine von [[Peptid]]en und [[Protein]]en (Eiweißen) sind. Bisher sind über zwanzig sogenannte ''proteinogene'' Aminosäuren bekannt. Dies sind zunächst jene 20 <small>L</small>-α-Aminosäuren, die als Standard-Aminosäuren durch [[Codon]]s von je drei [[Nukleinbasen]] in der [[Desoxyribonukleinsäure|DNA]] nach dem Standard-Code [[Genetischer Code|codiert]] werden. Zu diesen ''kanonisch'' genannten Aminosäuren sind inzwischen zwei weitere hinzugekommen, [[Selenocystein]] und [[Pyrrolysin]]. Beide nicht-kanonischen sind ebenfalls α-Aminosäuren, bezogen auf die endständige [[Carboxygruppe]] ist die [[Aminogruppe]] also am unmittelbar benachbarten Kohlenstoffatom gebunden (C<sub>α</sub>). Darüber hinaus gibt es noch weitere Aminosäuren, die als Bestandteil von Proteinen oder Peptiden auftreten, jedoch nicht codiert werden. |
<small>L</small>-Aminosäuren sind in der [[Biochemie]] von großer Bedeutung, da sie die Bausteine von [[Peptid]]en und [[Protein]]en (Eiweißen) sind. Bisher sind über zwanzig sogenannte ''proteinogene'' Aminosäuren bekannt. Dies sind zunächst jene 20 <small>L</small>-α-Aminosäuren, die als Standard-Aminosäuren durch [[Codon]]s von je drei [[Nukleinbasen]] in der [[Desoxyribonukleinsäure|DNA]] nach dem Standard-Code [[Genetischer Code|codiert]] werden. Zu diesen ''kanonisch'' genannten Aminosäuren sind inzwischen zwei weitere hinzugekommen, [[Selenocystein]] und [[Pyrrolysin]]. Beide nicht-kanonischen sind ebenfalls α-Aminosäuren, bezogen auf die endständige [[Carboxygruppe]] ist die [[Aminogruppe]] also am unmittelbar benachbarten Kohlenstoffatom gebunden (C<sub>α</sub>). Darüber hinaus gibt es noch weitere Aminosäuren, die als Bestandteil von Proteinen oder Peptiden auftreten, jedoch nicht codiert werden. |
||
| Zeile 245: | Zeile 232: | ||
=== Chemisch-physikalische Eigenschaften === |
=== Chemisch-physikalische Eigenschaften === |
||
[[Datei:Amino Acids Venn Diagram (de).svg|mini|hochkant= |
[[Datei:Amino Acids Venn Diagram (de).svg|mini|hochkant=0.9|Darstellung von Eigenschaften der Seitenketten proteinogener Standard-Aminosäuren im [[Mengendiagramm]]<ref name="Taylor" />]] |
||
Die proteinogenen Aminosäuren lassen sich nach ihren Resten in Gruppen aufteilen (siehe [[#Tabellenübersicht der Eigenschaften|Tabellenübersicht der Eigenschaften]]). Dabei kann eine Aminosäure in verschiedenen Gruppen gleichzeitig auftauchen. In einem [[Mengendiagramm]] lassen sich die Überlappungen der Gruppen grafisch darstellen. |
Die proteinogenen Aminosäuren lassen sich nach ihren Resten in Gruppen aufteilen (siehe [[#Tabellenübersicht der Eigenschaften|Tabellenübersicht der Eigenschaften]]). Dabei kann eine Aminosäure in verschiedenen Gruppen gleichzeitig auftauchen. In einem [[Mengendiagramm]] lassen sich die Überlappungen der Gruppen grafisch darstellen. |
||
| Zeile 251: | Zeile 238: | ||
==== Säure- und Basen-Verhalten ==== |
==== Säure- und Basen-Verhalten ==== |
||
Für das Säure-Base-Verhalten proteinogener Aminosäuren ist vor allem das Verhalten der Seitenkette der Aminosäure (fortan mit ''R'' bezeichnet) interessant. In Proteinen sind die NH<sub>2</sub>- und COOH-Gruppen bei physiologischen pH-Wert (um pH 7) wegen der [[Peptidbindung]] nicht protonierbar und damit auch nicht [[Säure-Base-Titration|titrierbar]]. Die Ausnahme ist der [[Amino-Terminus|Amino]]- und der [[Carboxy-Terminus]] des Proteins. Daher ist für das Säure-Base-Verhalten von Proteinen und Peptiden der Seitenkettenrest ''R'' maßgeblich. |
|||
Das Verhalten der Seitenkette ''R'' hängt von ihrer Konstitution ab, das heißt, ob die Seitenkette selbst wieder als [[Protonenakzeptor]] oder -[[Protonendonator|donator]] wirken kann. Die proteinogenen Aminosäuren werden nach den funktionellen Gruppen in unpolare oder polare Aminosäureseitenketten eingeteilt und weiter (in nach [[Polarität (Chemie)|Polarität]] sortierte Untergruppen) in ''aliphatische'', ''aromatische'', ''amidierte'', ''Schwefel-enthaltende'', ''hydroxylierte'', ''basische'' und ''saure'' Aminosäuren unterteilt. Die Seitenketten von Tyrosin und Cystein sind zwar im Vergleich zu den anderen unpolaren Seitenketten relativ sauer, neigen aber erst bei unphysiologisch hohen pH-Werten zum Deprotonieren. Prolin ist eine sekundäre Aminosäure, da der N-Terminus mit der Seitenkette einen fünfatomigen Ring schließt. Innerhalb eines Proteins bindet der Carboxy-Terminus einer vorhergehenden Aminosäure an den Stickstoff des Prolins, welcher aufgrund der bereits erwähnten Peptidbindung nicht protonierbar ist. Histidin, Tyrosin und Methionin kommen jeweils in zwei Untergruppen vor. |
|||
{| class="wikitable float-right" style="text-align:center" |
{| class="wikitable float-right" style="text-align:center" |
||
|+ pKa-Werte von Aminosäure-Seitenketten (für die freien Aminosäurenreste und im Protein) |
|+ pKa-Werte von Aminosäure-Seitenketten (für die freien Aminosäurenreste und im Protein) |
||
| Zeile 256: | Zeile 247: | ||
! Aminosäure !! Bezeichnung !! frei !! im Protein |
! Aminosäure !! Bezeichnung !! frei !! im Protein |
||
|- |
|- |
||
| [[Asparaginsäure|Asp]] || sauer || |
| [[Asparaginsäure|Asp]] || sauer || 3,68 ||3,7–4,0 |
||
|- |
|- |
||
| [[Glutaminsäure|Glu]] || sauer || |
| [[Glutaminsäure|Glu]] || sauer || 4,25 || 4,2–4,5 |
||
|- |
|- |
||
| [[Histidin|His]] || basisch || |
| [[Histidin|His]] || basisch || 6,0 || 6,7–7,1 |
||
|- |
|- |
||
| [[Cystein|Cys]] || |
| [[Cystein|Cys]] || semisauer || 8,33 || 8,8–9,1 |
||
|- |
|- |
||
| [[Tyrosin|Tyr]] || |
| [[Tyrosin|Tyr]] || semisauer || 10,07 || 9,7–10,1 |
||
|- |
|- |
||
| [[Lysin|Lys]] || basisch || 10,53 || |
| [[Lysin|Lys]] || basisch || 10,53 || 9,3–9,5 |
||
|- |
|- |
||
| [[Arginin|Arg]] || basisch || 12,48 || – |
| [[Arginin|Arg]] || basisch || 12,48 || – |
||
|} |
|} |
||
''Aliphatische Aminosäureseitenketten'' |
|||
Für das Säure-Base-Verhalten proteinogener Aminosäuren ist vor allem das Verhalten der Seitenkette der Aminosäure (fortan mit ''R'' bezeichnet) interessant. In Proteinen sind die NH<sub>2</sub>- und COOH-Gruppen bei physiologischen pH-Wert (um pH 7) wegen der [[Peptidbindung]] nicht protonierbar und damit auch nicht [[Säure-Base-Titration|titrierbar]]. Die Ausnahme ist der [[Amino-Terminus|Amino]]- und der [[Carboxy-Terminus]] des Proteins. Daher ist für das Säure-Base-Verhalten von Proteinen und Peptiden der Seitenkettenrest ''R'' maßgeblich. |
|||
* [[Alanin]] |
|||
* [[Glycin]] |
|||
* [[Isoleucin]] |
|||
* [[Leucin]] |
|||
* [[Methionin]] |
|||
* [[Prolin]] |
|||
* [[Valin]] |
|||
''Aromatische Aminosäureseitenketten'' |
|||
Das Verhalten der Seitenkette ''R'' hängt von ihrer Konstitution ab, das heißt, ob die Seitenkette selbst wieder als [[Protonenakzeptor]] oder -[[Protonendonator|donator]] wirken kann. Die proteinogenen Aminosäuren werden nach den funktionellen Gruppen in unpolare oder polare Aminosäureseitenketten eingeteilt und weiter (in nach [[Polarität (Chemie)|Polarität]] sortierte Untergruppen) in ''aliphatische'', ''aromatische'', ''amidierte'', ''Schwefel-enthaltende'', ''hydroxylierte'', ''basische'' und ''saure'' Aminosäuren unterteilt. Die Seitenketten von Tyrosin und Cystein sind zwar im Vergleich zu den anderen unpolaren Seitenketten relativ sauer, neigen aber erst bei unphysiologisch hohen pH-Werten zum Deprotonieren. Prolin ist eine sekundäre Aminosäure, da der N-Terminus mit der Seitenkette einen fünfatomigen Ring schließt. Innerhalb eines Proteins bindet der Carboxy-Terminus einer vorhergehenden Aminosäure an den Stickstoff des Prolins, welcher aufgrund der bereits erwähnten Peptidbindung nicht protonierbar ist. Histidin, Tyrosin und Methionin kommen jeweils in zwei Untergruppen vor. |
|||
* [[Histidin]] |
|||
* [[Phenylalanin]] |
|||
* [[Tryptophan]] |
|||
* [[Tyrosin]] |
|||
''Amidierte Aminosäureseitenketten'' |
|||
{| class="wikitable sortable float-right" style="text-align:center" |
|||
* [[Asparagin]] |
|||
* [[Glutamin]] |
|||
''Schwefel-enthaltende Aminosäureseitenketten'' |
|||
* [[Cystein]] |
|||
* [[Methionin]] |
|||
''Hydroxylierte Aminosäureseitenketten'' |
|||
* [[Serin]] |
|||
* [[Threonin]] |
|||
* [[Tyrosin]] |
|||
''Basische Aminosäureseitenketten'' |
|||
* [[Lysin]] |
|||
* [[Arginin]] |
|||
* [[Histidin]] |
|||
''Saure Aminosäureseitenketten'' |
|||
* [[Asparaginsäure]] (in Wasser dissoziiert zu Aspartat) |
|||
* [[Glutaminsäure]] (in Wasser dissoziiert zu Glutamat) |
|||
Der pK-Wert ist der [[pH-Wert]], bei dem die titrierbaren Gruppen zu gleichen Teilen protoniert und deprotoniert vorliegen. D. h., die titrierbare Gruppe liegt zu gleichen Teilen in ihrer basischen, wie in ihrer sauren Form vor (siehe auch: [[Henderson-Hasselbalch-Gleichung]]). |
|||
Es ist meist üblich, anstatt vom pK<sub>S</sub> vom pK zu sprechen, das heißt vom ''pK der Säure''. In diesem Sinne müsste allerdings vom pK des Lysins als pK<sub>B</sub>, also vom ''pK der Base'' gesprochen werden. Aus Gründen der Vereinfachung wird diese Notation aber allgemein weggelassen, da sie sich auch aus dem Sinnzusammenhang selbst ergibt (d. h. ob die Gruppe als Base oder Säure wirkt). |
|||
Der pK ist keine Konstante, sondern hängt von der Temperatur, der [[Aktivität (Chemie)|Aktivität]], der [[Ionenstärke]] und der unmittelbaren Umgebung der titrierbaren Gruppe ab und kann daher stark schwanken. |
|||
Ist der pH höher als der pK einer titrierbaren Gruppe, so liegt die titrierbare Gruppe in ihrer basischen (deprotonierten) Form vor. Ist der pH niedriger als der pK der titrierbaren Gruppe, so liegt die titrierbare Gruppe in ihrer sauren (protonierten) Form vor: |
|||
* Für Asp (pK = 3,86) bei pH 7: Die Seitenkette ist nahezu vollständig deprotoniert. |
|||
* Für Lys (pK= 10,53) bei pH 7: Die Seitenkette ist nahezu vollständig protoniert. |
|||
Die Seitenketten ''basischer Aminosäuren'' sind in ihrer protonierten (sauren) Form einfach positiv geladen und in ihrer deprotonierten (basischen) Form ungeladen. Die Seitenketten der ''sauren Aminosäuren'' (einschließlich Cystein und Tyrosin) sind in ihrer protonierten (sauren) Form ungeladen und in ihrer deprotonierten (basischen) Form einfach negativ geladen. Deswegen spielt der pH-Wert für die Eigenschaften der Seitenkette eine so wichtige Rolle, da das Verhalten der Seitenkette ein ganz anderes ist, wenn sie geladen bzw. ungeladen ist. |
|||
Die titrierbaren Seitenketten beeinflussen zum Beispiel das [[Löslichkeit]]sverhalten der entsprechenden Aminosäure. In polaren Lösungsmitteln gilt: Geladene Seitenketten machen die Aminosäure löslicher, ungeladene Seitenketten machen die Aminosäure unlöslicher. |
|||
In [[Proteine]]n kann das dazu führen, dass bestimmte Abschnitte [[Hydrophilie|hydrophiler]] oder [[Hydrophobie|hydrophober]] werden, wodurch die Faltung und damit die Aktivität von [[Enzym]]en vom pH-Wert abhängt. Durch stark saure oder basische Lösungen können Proteine deswegen auch [[Denaturierung (Biochemie)|denaturiert]] werden. |
|||
{| class="wikitable sortable" style="text-align:center" |
|||
|+ Elektrische Eigenschaften der Aminosäuren<ref name="Taylor" /> |
|+ Elektrische Eigenschaften der Aminosäuren<ref name="Taylor" /> |
||
|- class="hintergrundfarbe6" |
|- class="hintergrundfarbe6" |
||
! Aminosäuren |
|||
! Amino-<br>säuren |
|||
! pK<sub>2</sub |
! pK<sub>2</sub>COOH |
||
! pK<sub>1</sub |
! pK<sub>1</sub>COOH |
||
! |
! Isoelektrischer<br />Punkt |
||
! pK<sub>1</sub |
! pK<sub>1</sub>NH<sub>2</sub> |
||
! pK<sub>2</sub |
! pK<sub>2</sub>NH<sub>2</sub> |
||
|- |
|- |
||
| [[Alanin]] |
| [[Alanin]] |
||
| – |
| – |
||
| |
| 2,3 |
||
| |
| 6,1 |
||
| |
| 9,9 |
||
| – |
| – |
||
|- |
|- |
||
| [[Arginin]] |
| [[Arginin]] |
||
| – |
| – |
||
| |
| 2,81 |
||
| 10,76 |
| 10,76 |
||
| |
| 9,09 |
||
| 13,2 |
| 13,2 |
||
|- |
|- |
||
| [[Asparagin]] |
| [[Asparagin]] |
||
| – |
| – |
||
| |
| 2,02 |
||
| |
| 5,41 |
||
| |
| 8,80 |
||
| – |
| – |
||
|- |
|- |
||
| [[Asparaginsäure]] |
| [[Asparaginsäure]] |
||
| |
| 3,65 |
||
| |
| 1,88 |
||
| |
| 2,85 |
||
| |
| 9,60 |
||
| – |
| – |
||
|- |
|- |
||
| [[Cystein]] |
| [[Cystein]] |
||
| |
| 8,33* |
||
| |
| 1,71 |
||
| |
| 5,05 |
||
| 10,78 |
| 10,78 |
||
| – |
| – |
||
| Zeile 322: | Zeile 362: | ||
| [[Glutamin]] |
| [[Glutamin]] |
||
| – |
| – |
||
| |
| 2,17 |
||
| |
| 5,65 |
||
| |
| 9,13 |
||
| – |
| – |
||
|- |
|- |
||
| [[Glutaminsäure]] |
| [[Glutaminsäure]] |
||
| |
| 4,25 |
||
| |
| 2,19 |
||
| |
| 3,22 |
||
| |
| 9,67 |
||
| – |
| – |
||
|- |
|- |
||
| [[Glycin]] |
| [[Glycin]] |
||
| – |
| – |
||
| |
| 2,21 |
||
| |
| 5,97 |
||
| |
| 9,15 |
||
| – |
| – |
||
|- |
|- |
||
| [[Histidin]] |
| [[Histidin]] |
||
| – |
| – |
||
| |
| 1,78 |
||
| |
| 7,47 |
||
| |
| 8,97 |
||
| |
| 5,97 |
||
|- |
|- |
||
| [[Isoleucin]] |
| [[Isoleucin]] |
||
| – |
| – |
||
| |
| 2,32 |
||
| |
| 5,94 |
||
| |
| 9,76 |
||
| – |
| – |
||
|- |
|- |
||
| [[Leucin]] |
| [[Leucin]] |
||
| – |
| – |
||
| |
| 2,4 |
||
| |
| 5,98 |
||
| |
| 9,6 |
||
| – |
| – |
||
|- |
|- |
||
| [[Lysin]] |
| [[Lysin]] |
||
| – |
| – |
||
| |
| 2,20 |
||
| |
| 9,59 |
||
| |
| 8,90 |
||
| 10,28 |
| 10,28 |
||
|- |
|- |
||
| [[Methionin]] |
| [[Methionin]] |
||
| – |
| – |
||
| |
| 2,28 |
||
| |
| 5,74 |
||
| |
| 9,21 |
||
| – |
| – |
||
|- |
|- |
||
| [[Phenylalanin]] |
| [[Phenylalanin]] |
||
| – |
| – |
||
| |
| 2,58 |
||
| |
| 5,84 |
||
| |
| 9,24 |
||
| – |
| – |
||
|- |
|- |
||
| [[Prolin]] |
| [[Prolin]] |
||
| – |
| – |
||
| |
| 1,99 |
||
| |
| 6,3 |
||
| 10,60 |
| 10,60 |
||
| – |
| – |
||
| Zeile 392: | Zeile 432: | ||
| [[Serin]] |
| [[Serin]] |
||
| – |
| – |
||
| |
| 2,21 |
||
| |
| 5,68 |
||
| |
| 9,15 |
||
| – |
| – |
||
|- |
|- |
||
| [[Threonin]] |
| [[Threonin]] |
||
| – |
| – |
||
| |
| 2,10 |
||
| |
| 5,60 |
||
| |
| 9,12 |
||
| – |
| – |
||
|- |
|- |
||
| [[Tryptophan]] |
| [[Tryptophan]] |
||
| – |
| – |
||
| |
| 2,15 |
||
| |
| 5,64 |
||
| |
| 9,12 |
||
| – |
| – |
||
|- |
|- |
||
| [[Tyrosin]] |
| [[Tyrosin]] |
||
| 10,07** |
| 10,07** |
||
| |
| 2,20 |
||
| |
| 5,66 |
||
| |
| 9,11 |
||
| – |
| – |
||
|- |
|- |
||
| [[Valin]] |
| [[Valin]] |
||
| – |
| – |
||
| |
| 2,30 |
||
| |
| 5,96 |
||
| |
| 9,60 |
||
| – |
| – |
||
|- class="sortbottom" style="text-align:left" |
|- class="sortbottom" style="text-align:left" |
||
| colspan="6" | <small><sup>*</sup> [[Thiolgruppe]]<br /><sup>**</sup> [[phenol]]ische [[Hydroxygruppe]]</small> |
| colspan="6" | <small><sup>*</sup> [[Thiolgruppe]]<br /><sup>**</sup> [[phenol]]ische [[Hydroxygruppe]]</small> |
||
|} |
|} |
||
''Aliphatische Aminosäureseitenketten'' |
|||
* [[Alanin]] |
|||
* [[Glycin]] |
|||
* [[Isoleucin]] |
|||
* [[Leucin]] |
|||
* [[Methionin]] |
|||
* [[Prolin]] |
|||
* [[Valin]] |
|||
''Aromatische Aminosäureseitenketten'' |
|||
* [[Histidin]] |
|||
* [[Phenylalanin]] |
|||
* [[Tryptophan]] |
|||
* [[Tyrosin]] |
|||
''Amidierte Aminosäureseitenketten'' |
|||
* [[Asparagin]] |
|||
* [[Glutamin]] |
|||
''Schwefel-enthaltende Aminosäureseitenketten'' |
|||
* [[Cystein]] |
|||
* [[Methionin]] |
|||
''Hydroxylierte Aminosäureseitenketten'' |
|||
* [[Serin]] |
|||
* [[Threonin]] |
|||
* [[Tyrosin]] |
|||
''Basische Aminosäureseitenketten'' |
|||
* [[Lysin]] |
|||
* [[Arginin]] |
|||
* [[Histidin]] |
|||
''Saure Aminosäureseitenketten'' |
|||
* [[Asparaginsäure]] (in Wasser dissoziiert zu Aspartat) |
|||
* [[Glutaminsäure]] (in Wasser dissoziiert zu Glutamat) |
|||
Der pK-Wert ist der [[pH-Wert]], bei dem die titrierbaren Gruppen zu gleichen Teilen protoniert und deprotoniert vorliegen. D. h., die titrierbare Gruppe liegt zu gleichen Teilen in ihrer basischen, wie in ihrer sauren Form vor (siehe auch: [[Henderson-Hasselbalch-Gleichung]]). |
|||
Es ist meist üblich, anstatt vom pK<sub>S</sub> vom pK zu sprechen, das heißt vom ''pK der Säure''. In diesem Sinne müsste allerdings vom pK des Lysins als pK<sub>B</sub>, also vom ''pK der Base'' gesprochen werden. Aus Gründen der Vereinfachung wird diese Notation aber allgemein weggelassen, da sie sich auch aus dem Sinnzusammenhang selbst ergibt (d. h. ob die Gruppe als Base oder Säure wirkt). |
|||
Der pK ist keine Konstante, sondern hängt von der Temperatur, der [[Aktivität (Chemie)|Aktivität]], der [[Ionenstärke]] und der unmittelbaren Umgebung der titrierbaren Gruppe ab und kann daher stark schwanken. |
|||
Ist der pH höher als der pK einer titrierbaren Gruppe, so liegt die titrierbare Gruppe in ihrer basischen (deprotonierten) Form vor. Ist der pH niedriger als der pK der titrierbaren Gruppe, so liegt die titrierbare Gruppe in ihrer sauren (protonierten) Form vor: |
|||
* Für Asp (pK = 3,86) bei pH 7: Die Seitenkette ist nahezu vollständig deprotoniert. |
|||
* Für Lys (pK= 10,53) bei pH 7: Die Seitenkette ist nahezu vollständig protoniert. |
|||
Die Seitenketten ''basischer Aminosäuren'' sind in ihrer protonierten (sauren) Form einfach positiv geladen und in ihrer deprotonierten (basischen) Form ungeladen. Die Seitenketten der ''sauren Aminosäuren'' (einschließlich Cystein und Tyrosin) sind in ihrer protonierten (sauren) Form ungeladen und in ihrer deprotonierten (basischen) Form einfach negativ geladen. Deswegen spielt der pH-Wert für die Eigenschaften der Seitenkette eine so wichtige Rolle, da das Verhalten der Seitenkette ein ganz anderes ist, wenn sie geladen bzw. ungeladen ist. |
|||
Die titrierbaren Seitenketten beeinflussen zum Beispiel das [[Löslichkeit]]sverhalten der entsprechenden Aminosäure. In polaren Lösungsmitteln gilt: Geladene Seitenketten machen die Aminosäure löslicher, ungeladene Seitenketten machen die Aminosäure unlöslicher. |
|||
In [[Proteine]]n kann das dazu führen, dass bestimmte Abschnitte [[Hydrophilie|hydrophiler]] oder [[Hydrophobie|hydrophober]] werden, wodurch die Faltung und damit die Aktivität von [[Enzym]]en vom pH-Wert abhängt. Durch stark saure oder basische Lösungen können Proteine deswegen auch [[Denaturierung (Biochemie)|denaturiert]] werden. |
|||
==== Tabellenübersicht der Eigenschaften ==== |
==== Tabellenübersicht der Eigenschaften ==== |
||
{| class="wikitable sortable" style="text-align:center |
{| class="wikitable sortable" style="text-align:center" |
||
|+ Eigenschaften der 20 kanonischen Aminosäuren ('''R''' = Seitenkette) nach Taylor.<ref name="Taylor" /> |
|+ Eigenschaften der 20 kanonischen Aminosäuren ('''R''' = Seitenkette) nach Taylor.<ref name="Taylor" /> |
||
|- class="hintergrundfarbe6" |
|- class="hintergrundfarbe6" |
||
! Aminosäure |
|||
! rowspan="2" | Amino-<br>säure |
|||
! |
! Abk. |
||
! |
! Code |
||
! Aminosäure-Seitenkette ('''R''') |
|||
! rowspan="2" | Struktur-<br />formel<br>der<br>Aminosäure |
|||
! Charakter von '''R''' |
|||
! rowspan="2" | Aminosäure-<br>Seitenkette<br>R |
|||
! Molare Masse von '''R''' (g/mol) |
|||
! Charakter |
|||
! [[Van-der-Waals-Radius|van-der-Waals-Volumen]] von '''R''' |
|||
! Molare<br>Masse<br>(g/mol) |
|||
! [[Polarität (Chemie)|Polarität]] von '''R''' |
|||
! [[Van-der-Waals-Radius|van-der-<br>Waals-<br>Volumen]] |
|||
! [[Hydrophobizität|Hydro-<br />phobi-<br />zität]]<ref name="Kyte-Doolittle" /> von '''R''' |
|||
! [[Polarität (Chemie)|Pola-<br>rität]] |
|||
! [[Acidität]] oder [[Basizität]] von '''R''' |
|||
! [[Hydrophobizität|Hydro-<br />phobi-<br />zität]]<ref name="Kyte-Doolittle" /> |
|||
! [[Säurekonstante|Säure-Konstante]] (pKs)<br />von '''R''' |
|||
! [[Acidität]]<br>bzw.<br>[[Basizität]] |
|||
! Struktur-<br />formel |
|||
! [[Säurekonstante|Säure-<br>konstante]]<br>(pKs) |
|||
|- class="hintergrundfarbe6" |
|||
! colspan="7" | von '''R''' |
|||
|- |
|- |
||
| [[Alanin]] |
| [[Alanin]] |
||
| Ala |
| Ala |
||
| A |
| A |
||
| [[Datei:L-Alanin - L-Alanine.svg|rahmenlos|rechts|x45px|L-Alanin]] |
|||
| align="left" | –CH<sub>3</sub> |
| align="left" | –CH<sub>3</sub> |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 511: | Zeile 496: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Alanin - L-Alanine.svg|rahmenlos|zentriert|x40px|L-Alanin]] |
|||
|- |
|- |
||
| [[Arginin]] |
| [[Arginin]] |
||
| Arg |
| Arg |
||
| R |
| R |
||
|[[Datei:L-Arginin - L-Arginine.svg|rahmenlos|x45px|rechts|L-Arginin]] |
|||
| align="left" | –CH<sub>2</sub>CH<sub>2</sub>CH<sub>2</sub>[[Guanidin|NH-C(NH)NH<sub>2</sub>]] |
| align="left" | –CH<sub>2</sub>CH<sub>2</sub>CH<sub>2</sub>[[Guanidin|NH-C(NH)NH<sub>2</sub>]] |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 522: | Zeile 507: | ||
| polar |
| polar |
||
| −4,5 |
| −4,5 |
||
| basisch |
| basisch (stark) |
||
| 12,48 |
| 12,48 |
||
|[[Datei:L-Arginin - L-Arginine.svg|rahmenlos|zentriert|x40px|L-Arginin]] |
|||
|- |
|- |
||
| [[Asparagin]] |
| [[Asparagin]] |
||
| Asn |
| Asn |
||
| N |
| N |
||
|[[Datei:L-Asparagin - L-Asparagine.svg|rahmenlos|rechts|x45px|L-Asparagin]] |
|||
| align="left" | –CH<sub>2</sub>[[Carbonsäureamid|CONH<sub>2</sub>]] |
| align="left" | –CH<sub>2</sub>[[Carbonsäureamid|CONH<sub>2</sub>]] |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 537: | Zeile 522: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Asparagin - L-Asparagine.svg|rahmenlos|zentriert|x40px|L-Asparagin]] |
|||
|- |
|- |
||
| [[Asparaginsäure]] |
| [[Asparaginsäure]] |
||
| Asp |
| Asp |
||
| D |
| D |
||
|[[Datei:L-Asparaginsäure - L-Aspartic acid.svg|rahmenlos|rechts|x45px|L-Asparaginsäure]] |
|||
| align="left" | –CH<sub>2</sub>[[Carbonsäure|COOH]] |
| align="left" | –CH<sub>2</sub>[[Carbonsäure|COOH]] |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 550: | Zeile 535: | ||
| sauer |
| sauer |
||
| 3,90 |
| 3,90 |
||
|[[Datei:L-Asparaginsäure - L-Aspartic acid.svg|rahmenlos|zentriert|x40px|L-Asparaginsäure]] |
|||
|- |
|- |
||
| [[Cystein]] |
| [[Cystein]] |
||
| Cys |
| Cys |
||
| C |
| C |
||
|[[Datei:L-Cystein - L-Cysteine.svg|rahmenlos|rechts|x45px|L-Cystein]] |
|||
| align="left" | –CH<sub>2</sub>[[Thiolgruppe|SH]] |
| align="left" | –CH<sub>2</sub>[[Thiolgruppe|SH]] |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 563: | Zeile 548: | ||
| neutral |
| neutral |
||
| 8,18 |
| 8,18 |
||
|[[Datei:L-Cystein - L-Cysteine.svg|rahmenlos|zentriert|x40px|L-Cystein]] |
|||
|- |
|- |
||
| [[Glutamin]] |
| [[Glutamin]] |
||
| Gln |
| Gln |
||
| Q |
| Q |
||
|[[Datei:L-Glutamin - L-Glutamine.svg|rahmenlos|rechts|x45px|L-Glutamin]] |
|||
| align="left" | –CH<sub>2</sub>CH<sub>2</sub>[[Carbonsäureamid|CONH<sub>2</sub>]] |
| align="left" | –CH<sub>2</sub>CH<sub>2</sub>[[Carbonsäureamid|CONH<sub>2</sub>]] |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 576: | Zeile 561: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Glutamin - L-Glutamine.svg|rahmenlos|zentriert|x40px|L-Glutamin]] |
|||
|- |
|- |
||
| [[Glutaminsäure]] |
| [[Glutaminsäure]] |
||
| Glu |
| Glu |
||
| E |
| E |
||
|[[Datei:L-Glutaminsäure - L-Glutamic acid.svg|rahmenlos|rechts|x45px|L-Glutaminsäure]] |
|||
| align="left" | –CH<sub>2</sub>CH<sub>2</sub>[[Carbonsäure|COOH]] |
| align="left" | –CH<sub>2</sub>CH<sub>2</sub>[[Carbonsäure|COOH]] |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 589: | Zeile 574: | ||
| sauer |
| sauer |
||
| 4,07 |
| 4,07 |
||
|[[Datei:L-Glutaminsäure - L-Glutamic acid.svg|rahmenlos|zentriert|x40px|L-Glutaminsäure]] |
|||
|- |
|- |
||
| [[Glycin]] |
| [[Glycin]] |
||
| Gly |
| Gly |
||
| G |
| G |
||
|[[Datei:Glycin - Glycine.svg|rahmenlos|rechts|x45px|L-Glycin]] |
|||
| align="left" | –H |
| align="left" | –H |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 602: | Zeile 587: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:Glycin - Glycine.svg|rahmenlos|zentriert|x40px|L-Glycin]] |
|||
|- |
|- |
||
| [[Histidin]] |
| [[Histidin]] |
||
| His |
| His |
||
| H |
| H |
||
|[[Datei:L-Histidin - L-Histidine.svg|rahmenlos|rechts|x45px|L-Histidin]] |
|||
| align="left" | –CH<sub>2</sub>([[Imidazol|C<sub>3</sub>H<sub>3</sub>N<sub>2</sub>]]) |
| align="left" | –CH<sub>2</sub>([[Imidazol|C<sub>3</sub>H<sub>3</sub>N<sub>2</sub>]]) |
||
| aromatisch |
| aromatisch |
||
| Zeile 613: | Zeile 598: | ||
| polar |
| polar |
||
| −3,2 |
| −3,2 |
||
| basisch |
| basisch (schwach) |
||
| 6,04 |
| 6,04 |
||
|[[Datei:L-Histidin - L-Histidine.svg|rahmenlos|zentriert|x40px|L-Histidin]] |
|||
|- |
|- |
||
| [[Isoleucin]] |
| [[Isoleucin]] |
||
| Ile |
| Ile |
||
| I |
| I |
||
|[[Datei:L-Isoleucin - L-Isoleucine.svg|rahmenlos|rechts|x45px|L-Isoleucin]] |
|||
| align="left" | –CH(CH<sub>3</sub>)CH<sub>2</sub>CH<sub>3</sub> |
| align="left" | –CH(CH<sub>3</sub>)CH<sub>2</sub>CH<sub>3</sub> |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 628: | Zeile 613: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Isoleucin - L-Isoleucine.svg|rahmenlos|zentriert|x40px|L-Isoleucin]] |
|||
|- |
|- |
||
| [[Leucin]] |
| [[Leucin]] |
||
| Leu |
| Leu |
||
| L |
| L |
||
|[[Datei:L-Leucin - L-Leucine.svg|rahmenlos|rechts|x45px|L-leucin]] |
|||
| align="left" | –CH<sub>2</sub>CH(CH<sub>3</sub>)<sub>2</sub> |
| align="left" | –CH<sub>2</sub>CH(CH<sub>3</sub>)<sub>2</sub> |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 641: | Zeile 626: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Leucin - L-Leucine.svg|rahmenlos|zentriert|x40px|L-leucin]] |
|||
|- |
|- |
||
| [[Lysin]] |
| [[Lysin]] |
||
| Lys |
| Lys |
||
| K |
| K |
||
|[[Datei:L-Lysin - L-Lysine.svg|rahmenlos|rechts|x45px|L-Lysin]] |
|||
| align="left" | –CH<sub>2</sub>CH<sub>2</sub>CH<sub>2</sub>CH<sub>2</sub>[[Aminogruppe|NH<sub>2</sub>]] |
| align="left" | –CH<sub>2</sub>CH<sub>2</sub>CH<sub>2</sub>CH<sub>2</sub>[[Aminogruppe|NH<sub>2</sub>]] |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 654: | Zeile 639: | ||
| basisch |
| basisch |
||
| 10,54 |
| 10,54 |
||
|[[Datei:L-Lysin - L-Lysine.svg|rahmenlos|zentriert|x40px|L-Lysin]] |
|||
|- |
|- |
||
| [[Methionin]] |
| [[Methionin]] |
||
| Met |
| Met |
||
| M |
| M |
||
|[[Datei:L-Methionin - L-Methionine.svg|rahmenlos|rechts|x45px|L-Methionin]] |
|||
| align="left" | –CH<sub>2</sub>CH<sub>2</sub>[[Thioether|SCH<sub>3</sub>]] |
| align="left" | –CH<sub>2</sub>CH<sub>2</sub>[[Thioether|SCH<sub>3</sub>]] |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 667: | Zeile 652: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Methionin - L-Methionine.svg|rahmenlos|zentriert|x40px|L-Methionin]] |
|||
|- |
|- |
||
| [[Phenylalanin]] |
| [[Phenylalanin]] |
||
| Phe |
| Phe |
||
| F |
| F |
||
|[[Datei:L-Phenylalanin - L-Phenylalanine.svg|rahmenlos|rechts|x45px|L-Phenylalanin]] |
|||
| align="left" | –CH<sub>2</sub>([[Phenyl|C<sub>6</sub>H<sub>5</sub>]]) |
| align="left" | –CH<sub>2</sub>([[Phenyl|C<sub>6</sub>H<sub>5</sub>]]) |
||
| aromatisch |
| aromatisch |
||
| Zeile 680: | Zeile 665: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Phenylalanin - L-Phenylalanine.svg|rahmenlos|zentriert|x40px|L-Phenylalanin]] |
|||
|- |
|- |
||
| [[Prolin]] |
| [[Prolin]] |
||
| Pro |
| Pro |
||
| P |
| P |
||
|[[Datei:L-Prolin - L-Proline.svg|rahmenlos|rechts|x45px|L-Prolin]] |
|||
| align="left" | Anmerkung<ref>Darstellung nicht verfügbar, da bei Prolin am Peptid-Rückgrat ein H-Atom am Stickstoff weniger vorkommt (ein [[sekundäres Amin]]), weil die Seitenkette mit dem Stickstoffatom einen Ring bildet (–NHCH<sub>2</sub>CH<sub>2</sub>CH<sub>2</sub>–).</ref> |
| align="left" | Anmerkung<ref>Darstellung nicht verfügbar, da bei Prolin am Peptid-Rückgrat ein H-Atom am Stickstoff weniger vorkommt (ein [[sekundäres Amin]]), weil die Seitenkette mit dem Stickstoffatom einen Ring bildet (–NHCH<sub>2</sub>CH<sub>2</sub>CH<sub>2</sub>–).</ref> |
||
| heterocyclisch |
| heterocyclisch |
||
| Zeile 693: | Zeile 678: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Prolin - L-Proline.svg|rahmenlos|zentriert|x40px|L-Prolin]] |
|||
|- |
|- |
||
| [[Serin]] |
| [[Serin]] |
||
| Ser |
| Ser |
||
| S |
| S |
||
|[[Datei:L-Serin - L-Serine.svg|rahmenlos|rechts|x45px|L-Serin]] |
|||
| align="left" | –CH<sub>2</sub>[[Hydroxygruppe|OH]] |
| align="left" | –CH<sub>2</sub>[[Hydroxygruppe|OH]] |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 706: | Zeile 691: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Serin - L-Serine.svg|rahmenlos|zentriert|x40px|L-Serin]] |
|||
|- |
|- |
||
| [[Threonin]] |
| [[Threonin]] |
||
| Thr |
| Thr |
||
| T |
| T |
||
|[[Datei:L-Threonin - L-Threonine.svg|rahmenlos|rechts|x45px|L-Threonin]] |
|||
| align="left" | –CH(OH)CH<sub>3</sub> |
| align="left" | –CH(OH)CH<sub>3</sub> |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 719: | Zeile 704: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Threonin - L-Threonine.svg|rahmenlos|zentriert|x40px|L-Threonin]] |
|||
|- |
|- |
||
| [[Tryptophan]] |
| [[Tryptophan]] |
||
| Trp |
| Trp |
||
| W |
| W |
||
|[[Datei:L-Tryptophan - L-Tryptophan.svg|rahmenlos|rechts|x45px|L-Tryptophan]] |
|||
| align="left" | –CH<sub>2</sub>([[Indol|C<sub>8</sub>H<sub>6</sub>N]]) |
| align="left" | –CH<sub>2</sub>([[Indol|C<sub>8</sub>H<sub>6</sub>N]]) |
||
| aromatisch |
| aromatisch |
||
| Zeile 732: | Zeile 717: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Tryptophan - L-Tryptophan.svg|rahmenlos|zentriert|x40px|L-Tryptophan]] |
|||
|- |
|- |
||
| [[Tyrosin]] |
| [[Tyrosin]] |
||
| Tyr |
| Tyr |
||
| Y |
| Y |
||
|[[Datei:L-Tyrosin - L-Tyrosine.svg|rahmenlos|rechts|x45px|L-Tyrosin]] |
|||
| align="left" | –CH<sub>2</sub>([[Phenyl|C<sub>6</sub>H<sub>4</sub>]])[[Hydroxygruppe|OH]] |
| align="left" | –CH<sub>2</sub>([[Phenyl|C<sub>6</sub>H<sub>4</sub>]])[[Hydroxygruppe|OH]] |
||
| aromatisch |
| aromatisch |
||
| Zeile 745: | Zeile 730: | ||
| neutral |
| neutral |
||
| 10,46 |
| 10,46 |
||
|[[Datei:L-Tyrosin - L-Tyrosine.svg|rahmenlos|zentriert|x40px|L-Tyrosin]] |
|||
|- |
|- |
||
| [[Valin]] |
| [[Valin]] |
||
| Val |
| Val |
||
| V |
| V |
||
|[[Datei:L-Valin - L-Valine.svg|rahmenlos|rechts|x45px|L-Valin]] |
|||
| align="left" | –CH(CH<sub>3</sub>)<sub>2</sub> |
| align="left" | –CH(CH<sub>3</sub>)<sub>2</sub> |
||
| aliphatisch |
| aliphatisch |
||
| Zeile 758: | Zeile 743: | ||
| neutral |
| neutral |
||
| – |
| – |
||
|[[Datei:L-Valin - L-Valine.svg|rahmenlos|zentriert|x40px|L-Valin]] |
|||
|} |
|} |
||
| Zeile 771: | Zeile 757: | ||
Die meisten nichtproteinogenen Aminosäuren leiten sich von den proteinogenen ab, die <small>L</small>-α-Aminosäuren sind. Dennoch können dabei auch β-Aminosäuren ([[β-Alanin]]) oder γ-Aminosäuren ([[Γ-Aminobuttersäure|GABA]]) entstehen. |
Die meisten nichtproteinogenen Aminosäuren leiten sich von den proteinogenen ab, die <small>L</small>-α-Aminosäuren sind. Dennoch können dabei auch β-Aminosäuren ([[β-Alanin]]) oder γ-Aminosäuren ([[Γ-Aminobuttersäure|GABA]]) entstehen. |
||
[[Datei:(all-S)-endo-cis-2-Azabicyclo-3.3.0-octane 3-carboxylic acid Formula V.1.svg|mini| |
[[Datei:(all-S)-endo-cis-2-Azabicyclo-3.3.0-octane 3-carboxylic acid Formula V.1.svg|mini|hochkant=0.9|Die nichtproteinogene synthetische Aminosäure (all-''S'')-''endo''-''cis''-2-Azabicyclo-[3.3.0]-octan-3-carbonsäure, ein Strukturelement des [[Arzneistoff]]s [[Ramipril]].]] |
||
Zu den nichtproteinogenen Aminosäuren zählen auch alle <small>D</small>-[[Enantiomer]]e der proteinogenen <small>L</small>-Aminosäuren. <small>D</small>-Serin wird im Hirn durch die [[Serin]]-[[Racemase]] aus <small>L</small>-Serin (seinem [[Enantiomer]]) erzeugt. Es dient sowohl als [[Neurotransmitter]] als auch als [[Gliazelle|Glio]]<nowiki />transmitter durch die Aktivierung des [[NMDA-Rezeptor]]s, was zusammen mit Glutamat die Öffnung des Kanals erlaubt. Zum Öffnen des Ionenkanals muss Glutamat und entweder Glycin oder <small>D</small>-Serin binden. <small>D</small>-Serin ist an der Glycin-Bindungsstelle des [[Glutamatrezeptor]]s vom NMDA-Typ ein stärkerer [[Agonist (Pharmakologie)|Agonist]] als Glycin selbst, war aber zum Zeitpunkt der Erstbeschreibung der Glycin-Bindungsstelle noch unbekannt. <small>D</small>-Serin ist nach <small>D</small>-Aspartat die zweite <small>D</small>-Aminosäure, die in Menschen gefunden wurde.<ref>{{Cite journal | first1 = Jean-Pierre | last1 = Mothet | first2 = Angèle T. | last2 = Parent | first3 = Herman | last3 = Wolosker | first4 = Roscoe O., Jr. | last4 = Brady | first5 = David J. | last5 = Linden | first6 = Christopher D. | last6 = Ferris | first7 = Michael A. | last7 = Rogawski | first8 = Solomon H. | last8 = Snyder | title = d-Serine is an endogenous ligand for the glycine site of the ''N''-methyl-d-aspartate receptor | journal = Proc. Natl. Acad. Sci. USA | volume = 97 | issue = 9 | pages = 4926–4931 | year = 2000 | pmid = 10781100 | pmc = 18334 | doi = 10.1073/pnas.97.9.4926 }}</ref> |
Zu den nichtproteinogenen Aminosäuren zählen auch alle <small>D</small>-[[Enantiomer]]e der proteinogenen <small>L</small>-Aminosäuren. <small>D</small>-Serin wird im Hirn durch die [[Serin]]-[[Racemase]] aus <small>L</small>-Serin (seinem [[Enantiomer]]) erzeugt. Es dient sowohl als [[Neurotransmitter]] als auch als [[Gliazelle|Glio]]<nowiki />transmitter durch die Aktivierung des [[NMDA-Rezeptor]]s, was zusammen mit Glutamat die Öffnung des Kanals erlaubt. Zum Öffnen des Ionenkanals muss Glutamat und entweder Glycin oder <small>D</small>-Serin binden. <small>D</small>-Serin ist an der Glycin-Bindungsstelle des [[Glutamatrezeptor]]s vom NMDA-Typ ein stärkerer [[Agonist (Pharmakologie)|Agonist]] als Glycin selbst, war aber zum Zeitpunkt der Erstbeschreibung der Glycin-Bindungsstelle noch unbekannt. <small>D</small>-Serin ist nach <small>D</small>-Aspartat die zweite <small>D</small>-Aminosäure, die in Menschen gefunden wurde.<ref>{{Cite journal | first1 = Jean-Pierre | last1 = Mothet | first2 = Angèle T. | last2 = Parent | first3 = Herman | last3 = Wolosker | first4 = Roscoe O., Jr. | last4 = Brady | first5 = David J. | last5 = Linden | first6 = Christopher D. | last6 = Ferris | first7 = Michael A. | last7 = Rogawski | first8 = Solomon H. | last8 = Snyder | title = d-Serine is an endogenous ligand for the glycine site of the ''N''-methyl-d-aspartate receptor | journal = Proc. Natl. Acad. Sci. USA | volume = 97 | issue = 9 | pages = 4926–4931 | year = 2000 | pmid = 10781100 | pmc = 18334 | doi = 10.1073/pnas.97.9.4926 }}</ref> |
||
Version vom 2. April 2017, 00:13 Uhr
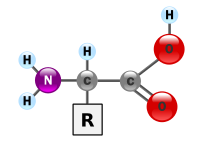
(im Fall von Glycin ist Rest R = H)
Aminosäuren (AS), unüblich auch Aminocarbonsäuren, veraltet Amidosäuren genannt, sind eine biologisch wichtige Klasse organischer Verbindungen, die als funktionelle Gruppen mindestens eine Carboxygruppe (–COOH) und eine Aminogruppe (–NH2 bzw. substituiert –NR2) enthalten, also Strukturmerkmale der Amine und der Carbonsäuren aufweisen. Aminosäuren werden hinsichtlich ihrer molekularen Grundstruktur chemisch in Klassen eingeteilt nach der Stellung der Aminogruppe zur Carboxygruppe. Steht die Aminogruppe am Cα-Atom in unmittelbarer Nachbarschaft zu der endständigen Carboxygruppe, nennt man dies α-ständig und spricht so von α-Aminosäuren.
Ausgewählte α-Aminosäuren sind die natürlichen Bausteine von Proteinen. Sie werden miteinander zu Ketten verknüpft, indem die Carboxygruppe der einen Aminosäure mit der Aminogruppe der nächsten eine Peptidbindung eingeht. Die auf diese Weise zu einem Polymer verketteten Aminosäuren unterscheiden sich in ihren Seitenketten und bestimmen zusammen die Form, mit der das Polypeptid im wässrigen Milieu dann zum nativen Protein auffaltet. Diese Biosynthese von Proteinen findet in allen Zellen an den Ribosomen statt nach Vorgabe genetischer Information, die in Form von mRNA vorliegt. Die hiermit als Bausteine für die Bildung von Proteinen codierten Aminosäuren werden auch als proteinogene Aminosäuren bezeichnet,[1] beim Menschen sind es 21 verschiedene. Nach der Translation können die Seitenketten einiger im Protein eingebauter Aminosäuren noch modifiziert werden.
Das Spektrum der Aminosäuren geht allerdings über die knapp zwei Dutzend proteinogenen weit hinaus. So sind bisher über 400 nichtproteinogene natürlich vorkommende Aminosäuren bekannt, die biologische Funktionen haben.[2] Die Anzahl der synthetisch erzeugten und die der theoretisch möglichen Aminosäuren ist noch erheblich größer. Eine spezielle Gruppe stellen die vergleichsweise seltenen D-Aminosäuren dar.
Abbauprodukte von Aminosäuren wie die biogenen Amine werden im Organismus vielfältig weiter umgesetzt und können selber physiologische Wirkungen, beispielsweise als Neurotransmitter, entfalten.
Aminosäuren konnten bisher nicht nur auf der Erde, sondern bereits auch auf Kometen, Meteoriten und sogar in Gaswolken im interstellaren Raum nachgewiesen werden.[3][4]
Geschichte
Die erste Aminosäure wurde 1805 im Pariser Labor von Louis-Nicolas Vauquelin und dessen Schüler Pierre Jean Robiquet aus dem Saft von Spargel (Asparagus officinalis) isoliert und danach Asparagin genannt.[5] 1931 wurde als letzte der üblichen proteinaufbauenden Aminosäuren das Threonin im Fibrin entdeckt und 1935 seiner Struktur nach geklärt von William Rose. Rose hatte durch Experimente mit verschiedenen Futtermitteln herausgefunden, dass die bis dato entdeckten 19 Aminosäuren als Zusatz nicht ausreichten.[6] Er stellte auch die Essentialität anderer Aminosäuren fest und ermittelte je die für ein optimales Wachstum mindestens erforderliche Tagesdosis.[7]
In der Zeit zwischen 1805 und 1935 waren viele der damals bekannten Chemiker und Pharmazeuten daran beteiligt, Aminosäuren erstmalig zu isolieren sowie deren Struktur aufzuklären. So gelang Emil Fischer, auf den auch die Fischer-Projektion zurückgeht, die finale Aufklärung der Struktur von Serin (1901), Lysin (1902), Valin (1906) und Cystein (1908). Auch Albrecht Kossel (1896 Histidin aus Störsperma), Richard Willstätter (1900 Prolin via Synthese) und Frederick Hopkins (1901 Tryptophan aus Casein) wurden später Nobelpreisträger. Der deutsche Chemiker Ernst Schulze isolierte drei Aminosäuren erstmals – 1877 Glutamin aus Rüben, 1881 Phenylalanin und 1886 Arginin aus Lupinen– und war an der Strukturaufklärung weiterer Aminosäuren beteiligt. Zuvor hatte Heinrich Ritthausen 1866 Glutaminsäure aus Getreideeiweiß, dem Gluten, kristallin gewonnen. Wilhelm Dittmar klärte 1872 die Struktur von Glutamin und Glutaminsäure, deren Salze Glutamate sind.

Bereits 1819 hatte Henri Braconnot das Glycin aus Leim und Joseph Louis Proust das Leucin aus Getreide isoliert. 1820 entdeckte William Hyde Wollaston das schwefelhaltige Cystin in Blasensteinen, doch erst 1884 Eugen Baumann das monomere Cystein. Eugen von Gorup-Besánez isolierte 1856 das Valin aus Pankreassaft. 1846 hatte Justus von Liebig aus Casein erstmals das Tyrosin abtrennen können, dessen Struktur 1869 Ludwig von Barth klärte. Im Hydrolysat des Casein entdeckte Edmund Drechsel 1889 auch das Lysin und später John Howard Mueller 1922 das schwefelhaltige Methionin als 19. Aminosäure, deren Strukturformel George Barger und Philip Coine 1928 angaben. In Melasse hatte Felix Ehrlich schon 1903 als 18. das Isoleucin gefunden, ein Strukturisomer des Leucin.
Friedrich Wöhler, dessen Synthesen in den 1820er Jahren das Gebiet der Biochemie eröffneten, entdeckte keine Aminosäure, doch waren drei seiner Schüler daran beteiligt, neben den erwähnten Gorup-Besánez und Schulze auch Georg Städeler (1863 Serin aus Rohseide). 18 der 20 entdeckten Aminosäuren wurden aus pflanzlichem oder tierischem Material isoliert, nur die beiden Aminosäuren Alanin (1850 Adolph Strecker) und Prolin (Willstätter) durch organische Synthese erhalten. Während die Analyse der stofflichen Zusammensetzung bis hin zur Summenformel mit den damaligen Methoden gut zu bewerkstelligen war, konnte die Strukturformel vieler Aminosäuren oftmals nur durch Teilschritte der Synthese endgültig aufgeklärt werden, was manchmal erst Jahre später gelang. Die Struktur des Asparagins beispielsweise – und die von Asparaginsäure – klärte Hermann Kolbe im Jahr 1862 auf, 57 Jahre nach der ersten Beschreibung.
Den Gattungsnamen verdanken Aminosäuren zwei funktionellen Gruppen, ihre Einzelnamen mal einem hellen Aussehen (z. B. Arginin, Leucin), einem süßen Geschmack (z. B. Glycin), dem Material, in dem sie gefunden wurden (z. B. Asparagin, Cystein, Serin, Tyrosin) Merkmalen der chemischen Struktur (z. B. Prolin, Valin, Isoleucin) oder beidem (z. B. Glutamin, Glutaminsäure) oder auch den Edukten ihrer Synthese (z. B. Alanin).[8]
Gewinnung und Produktion
Aminosäuren werden entweder aus Naturstoffen durch Auftrennung eines hydrolysierten Proteins oder auf synthetischem Wege gewonnen. Ursprünglich[9] diente die Entwicklung einer Synthese für die diversen Aminosäuren hauptsächlich der Strukturaufklärung. Inzwischen sind diese Strukturfragen gelöst und mit den verschiedenen Synthesen, soweit sie noch aktuell sind, werden gezielt die gewünschten Aminosäuren dargestellt. Bei den Synthesen entstehen zunächst racemische Gemische, die getrennt werden können. Eine Methode hierfür ist beispielsweise eine selektive enzymatische Hydrolyse. Näheres hierzu unter Trennverfahren.
Nachfolgend ein Überblick über diverse Synthesen, die von Chemikern bereits ab Mitte des 19. Jahrhunderts entwickelt wurden. Einige dieser älteren Synthesen sind wegen geringer Ausbeuten oder sonstiger Probleme nur von historischem Interesse. Allerdings wurden diese alten Verfahren teilweise weiterentwickelt und einige sind auch noch heute zur Darstellung von Aminosäuren aktuell. Weitergehende Einzelheiten zu diesen Synthesen einschließlich der Gleichungen für die Synthesen sind unter den Links zu den Synthesen und den angegebenen Aminosäuren angeführt.
- Mit der Cyanhydrinsynthese nach Adolf Strecker[10] wurde 1850 das Alanin erstmals aus Acetaldehyd synthetisiert.
- Eine Synthese für die Darstellung von Glycin über die α-Fettsäuren, die durch Reaktion von Brom- oder Chlorfettsäuren mit Ammoniak hergestellt werden, ist von W. H. Perkin sen. und Duppa bereits 1859[9] entwickelt worden.
- J. Pöchl entdeckte 1883 die Azlactonsynthese zur Darstellung von Aminosäuren. Deren genauer Ablauf wurde aber erst 1893 von Emil Erlenmeyer jun. aufgeklärt. Diese Methode wird deshalb auch Erlenmeyer-Synthese genannt. Mit diesem Verfahren wurden 1911[11] Histidin sowie Phenylalanin und Thyroxin hergestellt.
- Durch Reduktion von einer α-Oximinosäure wurde erstmals 1887[12] Asparaginsäure synthetisiert. Nach der gleichen Methode wurde 1906 von L. Bouveault Isoleucin aus dem Oxim des Methyläthyl-brenztraubensäureesters dargestellt.
- Nach der von Siegmund Gabriel entwickelten Gabriel-Synthese,[9] wurde 1889 Glycinhydrochlorid über Phthalimidkalium als Ausgangschemikalie synthetisiert. Obwohl diese Synthese für die Darstellung von Glycin überholt ist, eignet sie sich wegen ihrer hohen Ausbeuten für die Gewinnung anderer Aminosäuren.
- Mit der Cyanhydrinsynthese stellte Emil Fischer 1902 erstmals Serin[10] über Glykolaldehyd her. 1906 wurde mit der von ihm entwickelten Malonestersynthese[13] Leucin synthetisiert. Isoleucin, Norleucin, Methionin und Phenylalanin sind weitere Aminosäuren, die mit dieser Synthese leicht darstellbar sind.
- Theodor Curtius benutzte den von ihm entwickelten Curtiusschen Abbau für die Darstellung von α-Aminosäuren durch die Verwendung von Malonesterderivaten zur Synthese von Glycin, Alanin, Valin und Phenylalanin.[14]
- 1911[14] wurde Tyrosin, Phenylalanin und Tryptophan über eine Kondensation aromatischer Aldehyde mit Hydantoin gewonnen.
- Mit einer kombinierten Phthalimid-Malonester-Synthese wurde 1931[13] von G. Barger Methionin synthetisiert. Nach der gleichen Methode können auch Phenylalanin, Prolin, Tyrosin, Asparaginsäure und Serin hergestellt werden. V. du Vigneaud stellte 1939 DL-Cystin mit dieser Methode her.
Industriell werden Aminosäuren heute nach folgenden Verfahren hergestellt:
- Extraktionsmethode: Hierzu werden Proteine zunächst mit Säuren hydrolysiert. Nach Fällung des Aminosäuregemischs aus dem Hydrolysat erfolgt eine Trennung chromatographisch mit Hilfe von Ionenaustauschern, wobei bei der Elution die unterschiedlichen Polaritäten der Aminosäuren ausgenutzt werden.[15]
- Chemische Synthese: Es gibt eine Vielzahl von Synthesemethoden. Beispiele sind die Strecker-Synthese von D,L-Valin, die Degussa-Synthese von D,L-Cystein und die Synthese von D,L-Methionin aus Methylmercaptan, Acrolein und Blausäure. Da die hergestellten Aminosäuren dabei als Racemat erhalten werden, müssen anschließend noch Verfahren zur Enantiomerentrennung erfolgen, wenn reine L- oder D-Aminosäuren benötigt werden.
- Enzymatische Verfahren: Dieses Verfahren hat den Vorteil enantiomerenreine L- oder D-Aminosäuren mit geeigneten Enzymen als Biokatalysatoren zu liefern. Beispiele sind die Herstellung von L-Asparaginsäure aus Fumarsäure mit L-Aspartase und die Herstellung von L-Tryptophan aus Indol und Brenztraubensäure mit Tryptopharase.
- Fermentationsverfahren: Bei der Fermentation werden die Aminosäuren mit Hilfe geeigneter Mikroorganismen hergestellt. Der Syntheseprozess läuft dabei über sehr komplexe Zwischenschritte innerhalb der Zellen ab. Ein Beispiel ist die Herstellung von L-Glutaminsäure aus Glucose. Hierbei kann man aus 1 kg Glucose 0,5 kg Glutaminsäure gewinnen.
Aufbau der Aminosäuren
Aminosäuren bestehen aus mindestens zwei Kohlenstoffatomen. Die instabile Carbamidsäure besitzt lediglich ein Kohlenstoffatom und ist damit keine Aminosäure, sondern ein Kohlensäureamid. Aminosäuren lassen sich in Klassen einteilen je nach dem Kohlenstoffatom, an dem sich die Aminogruppe relativ zur Carboxygruppe befindet. Sind im Molekül mehrere Aminogruppen vertreten, so bestimmt das Kohlenstoffatom, dessen Aminogruppe dem Carboxy-Kohlenstoff am nächsten steht, um welche Klasse von Aminosäuren es sich handelt.
| Allgemeine Struktur von Aminosäuren (R = Seitenkette) |
 |
 |
 |
- α-Aminosäuren: Die Aminogruppe der α-Aminosäuren befindet sich am zweiten Kohlenstoffatom, einschließlich des Carboxy-Kohlenstoffatoms. Die Zählung beginnt immer mit dem Carboxy-Kohlenstoff. Die IUPAC-Bezeichnung lautet daher 2-Aminocarbonsäuren. Der einfachste Vertreter der α-Aminosäuren ist die proteinogene Aminosäure Glycin.
Der Begriff Aminosäuren wird meist synonym für eine Gruppe von α-Aminosäuren verwendet, die hauptsächlich aus L-α-Aminosäuren besteht: die proteinogenen Aminosäuren. Die proteinogenen Aminosäuren sind die Bausteine sämtlicher Proteine allen Lebens auf der Erde und neben den Nukleinsäuren Grundbausteine des Lebens. - β-Aminosäuren: Die Aminogruppe der β-Aminosäuren befindet sich am dritten Kohlenstoffatom (das Carboxy-Kohlenstoffatom mitgezählt). Die IUPAC-Bezeichnung lautet 3-Aminocarbonsäuren. Der einfachste Vertreter ist β-Alanin.
- γ-Aminosäuren: Die Aminogruppe der γ-Aminosäuren befindet sich am vierten Kohlenstoffatom (das Carboxy-Kohlenstoffatom mitgezählt). Die IUPAC-Bezeichnung lautet 4-Aminocarbonsäuren. Der einfachste Vertreter ist γ-Aminobuttersäure (GABA).
Die Bezeichnung weiterer Klassen der Aminosäuren ergibt sich nach dem gleichen Schema.
Die Aminosäuren einer Klasse unterscheiden sich durch ihre Seitenkette R. Ist die Seitenkette R verschieden von den anderen Substituenten, die sich am Kohlenstoff mit der Amino-Gruppe befinden, so befindet sich hier ein Stereozentrum und es existieren von der entsprechenden Aminosäure zwei Enantiomere. Enthält die Seitenkette R selbst weitere Stereozentren, so ergeben sich auch Diastereomere und die Zahl möglicher Stereoisomerer nimmt entsprechend zur Anzahl der weiteren Stereozentren zu. Von Aminosäuren mit zwei verschieden substituierten Stereozentren gibt es vier Stereoisomere.
Säure- und Base-Verhalten

Als Feststoffe und in neutralen wässrigen Lösungen liegen Aminosäuren als Zwitterionen vor, das heißt die Aminogruppe ist protoniert und die Carboxygruppe ist deprotoniert. Verallgemeinert lässt sich das Zwitterion so darstellen:
Als Zwitterion kann die protonierte Aminogruppe als Säure (Protonendonator) und die Carboxylatgruppe kann als Base (Protonenakzeptor) reagieren. In sauren Lösungen liegen Aminosäuren als Kationen und in basischen Lösungen als Anionen vor:
Die Ladung eines Aminosäuremoleküls hängt also vom pH-Wert der Lösung ab. Bei einem Zwitterion ist die Gesamtladung des Moleküls gleich null. Der entsprechende pH-Wert wird als isoelektrischer Punkt (pHI, pI) einer Aminosäure bezeichnet. Am isoelektrischen Punkt ist die Wasserlöslichkeit einer Aminosäure am geringsten.[16]
Proteinogene Aminosäuren
Als proteinogen werden Aminosäuren bezeichnet, die in Lebewesen als Bausteine der Proteine während der Translation nach Vorgabe genetischer Information verwendet werden.[1] Bei der Biosynthese von Proteinen, die an den Ribosomen einer Zelle stattfindet, werden ausgewählte Aminosäuren durch Peptidbindungen in bestimmter Reihenfolge zur Polypeptidkette eines Proteins verknüpft (→ Proteinbiosynthese). Die Aminosäurensequenz des ribosomal gebildeten Peptids wird dabei vorgegeben durch die in der Basensequenz einer Nukleinsäure enthaltene genetische Information, wobei eine Aminosäure durch ein Basentriplett codiert wird (→ genetischer Code).
 Proteinogene Aminosäure: Proteinogene Aminosäure: L-Prolin |
 Nichtproteinogene Aminosäure: Nichtproteinogene Aminosäure: D-Prolin |
Die proteinogenen Aminosäuren sind stets α-Aminosäuren. Bis auf die kleinste, Glycin, sind sie chiral und treten mit besonderer räumlicher Anordnung auf.[17] Von den spiegelbildlich verschiedenen Enantiomeren sind jeweils nur die L-Aminosäuren proteinogen (zur D / L-Nomenklatur siehe Fischer-Projektion; in Fällen wie Hydroxyprolin gibt es weitere Stereoisomere). Denn die molekularen Komponenten des zum Aufbau der Proteine notwendigen zellulären Apparats – neben Ribosomen noch tRNAs und diese mit Aminosäuren beladende Aminoacyl-tRNA-Synthetasen – sind selbst auch chiral und erkennen allein die L-Variante.[18]
Dennoch kommen in Lebewesen vereinzelt auch D-Aminosäuren vor. Diese werden jedoch unabhängig von proteinogenen Stoffwechselwegen synthetisiert und dienen nicht dem ribosomalen Aufbau von Proteinen. So wird zum Beispiel D-Alanin in Peptidoglycane der bakteriellen Zellwand eingebaut oder D-Valin in bakterielle Cyclo-Depsipeptide wie Valinomycin.
Kanonische Aminosäuren
Für 20 der proteinogenen Aminosäuren finden sich Codons in der (am häufigsten gebrauchten) Standardversion des genetischen Codes. Diese werden daher als Standardaminosäuren oder auch kanonische Aminosäuren bezeichnet.
Zu den Standardaminosäuren gehört die α-Aminosäure L-Prolin, die im Unterschied zu den übrigen kanonischen aber keine primäre, sondern eine sekundäre Aminogruppe besitzt und daher auch als sekundäre Aminosäure – öfters fälschlicherweise bzw. veraltet auch als Iminosäure – bezeichnet wird.
In Aminosäuresequenzen werden die Aminosäuren meist im Einbuchstabencode oder im Dreibuchstabencode dargestellt.[19]
| Aminosäure | Dreibuchstabencode | Einbuchstabencode | Acyl-Gruppe[19] | Bemerkung | Ø in Proteinen[20] |
|---|---|---|---|---|---|
| Alanin | Ala | A | Alanyl- | nicht-essentiell | 9,0 % |
| Arginin | Arg | R | Arginyl- | semi-essentiell | 4,7 % |
| Asparagin | Asn | N | Asparaginyl- | nicht-essentiell | 4,4 % |
| Asparaginsäure | Asp | D | α-Aspartyl- | nicht-essentiell | 5,5 % |
| Cystein | Cys | C | Cysteinyl- | nicht-essentiell* | 2,8 % |
| Glutamin | Gln | Q | Glutaminyl- | nicht-essentiell | 3,9 % |
| Glutaminsäure | Glu | E | α-Glutamyl- | nicht-essentiell | 6,2 % |
| Glycin | Gly | G | Glycyl- | nicht-essentiell | 7,5 % |
| Histidin | His | H | Histidyl- | semi-essentiell | 2,1 % |
| Isoleucin | Ile | I | Isoleucyl- | essentiell | 4,6 % |
| Leucin | Leu | L | Leucyl- | essentiell | 7,5 % |
| Lysin | Lys | K | Lysyl- | essentiell | 7,0 % |
| Methionin | Met | M | Methionyl- | essentiell | 1,7 % |
| Phenylalanin | Phe | F | Phenylalanyl- | essentiell | 3,5 % |
| Prolin | Pro | P | Prolyl- | nicht-essentiell | 4,6 % |
| Serin | Ser | S | Seryl- | nicht-essentiell | 7,1 % |
| Threonin | Thr | T | Threonyl- | essentiell | 6,0 % |
| Tryptophan | Trp | W | Tryptophyl- | essentiell | 1,1 % |
| Tyrosin | Tyr | Y | Tyrosyl- | nicht-essentiell* | 3,5 % |
| Valin | Val | V | Valyl- | essentiell | 6,9 % |
| *Für Kinder und Schwangere essentiell. | |||||
Neben den oben angegebenen Codes werden zusätzliche Zeichen als Platzhalter benutzt, wenn aus der Proteinsequenzierung oder Röntgenstrukturanalyse nicht auf die genaue Aminosäure geschlossen werden kann.
| Platzhalter | Dreibuchstabencode | Einbuchstabencode |
|---|---|---|
| Asparagin oder Asparaginsäure | Asx | B |
| Glutamin oder Glutaminsäure | Glx | Z |
| Leucin oder Isoleucin | Xle | J |
| unbekannte Aminosäure | Xaa (selten Unk) | X |
Nichtkanonische Aminosäuren
Zu den natürlich vorkommenden Aminosäuren gehören außer den kanonischen die übrigen als nichtkanonische Aminosäuren, wozu proteinogene und nicht-proteinogene zählen. Hierbei lassen sich mehrere Gruppen unterscheiden:


- Zur ersten Gruppe gehören jene proteinogenen Aminosäuren, die durch eine Recodierung des genetischen Materials in Proteine eingebaut werden. Die 21. und die 22. proteinogene Aminosäure – Selenocystein und Pyrrolysin – gehören hierzu. Für beide Aminosäuren wurden spezifische tRNAs – tRNASec bzw. tRNAPyl – gefunden, mit denen die Aminosäure jeweils für den ko-translationalen Einbau am Ribosom bereitgehalten wird. Deren Anticodon paart, abhängig von Strukturelementen im Kontext der mRNA, mit dem Codon UGA beziehungsweise UAG; im Standardcode stellen diese ein Stopcodon dar. Doch nicht alle Organismen verwenden die nichtkanonischen proteinogenen Aminosäuren dieser Gruppe.
| Aminosäure | Dreibuchstabencode | Einbuchstabencode |
|---|---|---|
| Pyrrolysin | Pyl | O |
| Selenocystein | Sec | U |

- Das übliche Startcodon AUG codiert für die Aminosäure Methionin. Bakterien verfügen neben der tRNAMet über eine besondere tRNAfMet, die ebenfalls mit Methionin beladen wird und als Initiator-tRNA dient. Die an tRNAifMet gebundene Aminosäure aber wird am N-Terminus formyliert zu N-Formylmethionin (fMet), noch bevor sie bei der Initation am Ribosom zur ersten Aminosäure einer Peptidkette werden kann. Dieses Aminosäurederivat Formylmethionin wird daher gelegentlich auch als (23.) proteinogene Aminosäure gezählt. Auch Mitochondrien und Chloroplasten nutzen fMet initial. Dagegen wird es im Cytosol eukaryotischer Zellen und in Archaeen nicht bei der Translation verwendet.[21]
- Eine zweite Gruppe bilden die im engen Sinn nicht proteinogenen Aminosäuren, die aus kanonischen Aminosäuren entstehen, wenn der Aminosäurerest R nach dem Einbau in Proteine verändert wird, also durch eine posttranslationale Modifikation. So kann beispielsweise Prolin zu Hydroxyprolin, Serin zu O-Phosphoserin, Tyrosin zu O-Phosphotyrosin und Glutamat zu γ-Carboxyglutamat umgewandelt werden. Eine wichtige Änderung des Aminosäurerestes stellt auch die Glykosylierung dar: Hier werden Kohlenhydratreste auf die Aminosäurereste übertragen, wodurch Glykoproteine entstehen.
- Als dritte Gruppe lassen sich die strenggenommen nicht proteinogenen Aminosäuren fassen, die der Organismus nicht von den kanonischen Aminosäuren unterscheiden kann und die er so anstelle dieser in Proteine unspezifisch einbaut. Dazu gehört beispielsweise Selenomethionin, das anstelle des Methionin eingebaut werden kann, oder das Canavanin, das der Organismus nicht vom Arginin unterscheiden kann oder auch die Azetidin-2-carbonsäure, die als Prolin-Analogon wirkt. Viele der Aminosäuren dieser Gruppe sind toxisch, da sie oft zu einer Fehlfaltung des Proteins führen, wodurch die Funktionsfähigkeit des Proteins beeinträchtigt werden kann. So ist Azetidin-2-carbonsäure ein toxischer Bestandteil des Maiglöckchens, wobei sich das Maiglöckchen selber mit einer hochspezifischen Prolyl-tRNA-Synthetase vor dem unkontrollierten Einbau dieser Aminosäure schützt.
Der Mensch nutzt neben den 20 kanonischen auch Selenocystein als proteinogene Aminosäure. Von den 20 kanonischen Aminosäuren werden 12 vom menschlichen Organismus beziehungsweise durch im menschlichen Verdauungstrakt lebende Mikroorganismen synthetisiert. Die restlichen 8 Aminosäuren sind für den Menschen essentiell, das heißt, er muss sie über die Nahrung aufnehmen.
Der Einbau künstlicher, nahezu beliebig gebauter Aminosäuren ist über die Ersetzung des Liganden in der entsprechenden Aminoacyl-tRNA-Synthetase möglich. Diese biotechnologischen Verfahren sind so weit fortgeschritten, dass damit gezielt bestimmte Proteine eine Markierung erhalten können, die das Protein nach Behandlung mit spezifischen Reagenzien zur Fluoreszenz anregen (Beispiel: Einbau von Norbornen-Aminosäure via Pyrrolysyl-tRNA-Synthetase/Codon CUA). Damit ist eine genaue Lokalisierung des Proteins auch ohne Produktion und Reaktion mit Antikörpern möglich.[22]
Biochemische Bedeutung
Aminosäuren als Bausteine von Proteinen

L-Aminosäuren sind in der Biochemie von großer Bedeutung, da sie die Bausteine von Peptiden und Proteinen (Eiweißen) sind. Bisher sind über zwanzig sogenannte proteinogene Aminosäuren bekannt. Dies sind zunächst jene 20 L-α-Aminosäuren, die als Standard-Aminosäuren durch Codons von je drei Nukleinbasen in der DNA nach dem Standard-Code codiert werden. Zu diesen kanonisch genannten Aminosäuren sind inzwischen zwei weitere hinzugekommen, Selenocystein und Pyrrolysin. Beide nicht-kanonischen sind ebenfalls α-Aminosäuren, bezogen auf die endständige Carboxygruppe ist die Aminogruppe also am unmittelbar benachbarten Kohlenstoffatom gebunden (Cα). Darüber hinaus gibt es noch weitere Aminosäuren, die als Bestandteil von Proteinen oder Peptiden auftreten, jedoch nicht codiert werden.
Aminosäureketten mit einer Kettenlänge unter zirka 100 Aminosäuren werden meist als Peptide bezeichnet, bei den größeren ribosomal gebildeten spricht man von Proteinen. Die einzelnen Aminosäuren sind dabei innerhalb der Kette je über Peptidbindungen (Säureamid) verknüpft. Ein automatisiertes Verfahren zur Synthese von Peptiden liefert die Merrifield-Synthese.
In Form von Nahrung aufgenommene Proteine werden bei der Verdauung in L-Aminosäuren zerlegt. In der Leber werden sie weiter verwertet. Entweder werden sie zur Proteinbiosynthese verwendet oder abgebaut (siehe auch: Aminosäureindex). Die wichtigsten Mechanismen des Aminosäurenabbaus sind:
Essentielle Aminosäuren
Aminosäuren, die ein Organismus benötigt, jedoch nicht selbst herstellen kann, heißen essentielle Aminosäuren und müssen mit der Nahrung aufgenommen werden. Alle diese essentiellen Aminosäuren sind L-α-Aminosäuren. Für Menschen sind Valin, Methionin, Leucin, Isoleucin, Phenylalanin, Tryptophan, Threonin und Lysin essentielle Aminosäuren. Bedingt essentielle oder semi-essentielle Aminosäuren müssen nur in bestimmten Situationen mit der Nahrung aufgenommen werden, zum Beispiel während des Wachstums oder nach schweren Verletzungen. Die übrigen Aminosäuren werden entweder direkt synthetisiert oder aus anderen Aminosäuren durch Modifikation gewonnen. So kann Cystein aus der essentiellen Aminosäure Methionin synthetisiert werden. Solange das Vermögen, aus Phenylalanin die Aminosäure Tyrosin herzustellen, noch nicht ausgereift ist, zählt auch diese neben den anderen zu den essentiellen Aminosäuren im Kindesalter. Aus ähnlichem Grund muss auch bei einer Phenylketonurie Tyrosin zugeführt werden. Daneben gibt es andere Erkrankungen, die den Aminosäurestoffwechsel beeinträchtigen und die Aufnahme einer eigentlich nicht-essentiellen Aminosäure unter Umständen erfordern. Alle essentiellen bzw. semi-essentiellen Aminosäuren, die der menschliche Körper benötigt, sind beispielsweise in Hühnereiern enthalten.
Pflanzen und Mikroorganismen können alle für sie notwendigen Aminosäuren selbst synthetisieren. Daher gibt es für sie keine essentiellen Aminosäuren.[17]
Chemisch-physikalische Eigenschaften

Die proteinogenen Aminosäuren lassen sich nach ihren Resten in Gruppen aufteilen (siehe Tabellenübersicht der Eigenschaften). Dabei kann eine Aminosäure in verschiedenen Gruppen gleichzeitig auftauchen. In einem Mengendiagramm lassen sich die Überlappungen der Gruppen grafisch darstellen.
Die Eigenschaften der Seitenkette von Cystein betreffend haben die Autoren unterschiedliche Ansichten: Löffler[24] hält sie für polar, während Alberts[25] sie für unpolar hält. Richtigerweise handelt es sich bei Schwefel um ein Heteroatom, folglich gilt: Die Seitenkette von Cystein hat schwach polare Eigenschaften.
Säure- und Basen-Verhalten
Für das Säure-Base-Verhalten proteinogener Aminosäuren ist vor allem das Verhalten der Seitenkette der Aminosäure (fortan mit R bezeichnet) interessant. In Proteinen sind die NH2- und COOH-Gruppen bei physiologischen pH-Wert (um pH 7) wegen der Peptidbindung nicht protonierbar und damit auch nicht titrierbar. Die Ausnahme ist der Amino- und der Carboxy-Terminus des Proteins. Daher ist für das Säure-Base-Verhalten von Proteinen und Peptiden der Seitenkettenrest R maßgeblich.
Das Verhalten der Seitenkette R hängt von ihrer Konstitution ab, das heißt, ob die Seitenkette selbst wieder als Protonenakzeptor oder -donator wirken kann. Die proteinogenen Aminosäuren werden nach den funktionellen Gruppen in unpolare oder polare Aminosäureseitenketten eingeteilt und weiter (in nach Polarität sortierte Untergruppen) in aliphatische, aromatische, amidierte, Schwefel-enthaltende, hydroxylierte, basische und saure Aminosäuren unterteilt. Die Seitenketten von Tyrosin und Cystein sind zwar im Vergleich zu den anderen unpolaren Seitenketten relativ sauer, neigen aber erst bei unphysiologisch hohen pH-Werten zum Deprotonieren. Prolin ist eine sekundäre Aminosäure, da der N-Terminus mit der Seitenkette einen fünfatomigen Ring schließt. Innerhalb eines Proteins bindet der Carboxy-Terminus einer vorhergehenden Aminosäure an den Stickstoff des Prolins, welcher aufgrund der bereits erwähnten Peptidbindung nicht protonierbar ist. Histidin, Tyrosin und Methionin kommen jeweils in zwei Untergruppen vor.
| Aminosäure | Bezeichnung | frei | im Protein |
|---|---|---|---|
| Asp | sauer | 3,68 | 3,7–4,0 |
| Glu | sauer | 4,25 | 4,2–4,5 |
| His | basisch | 6,0 | 6,7–7,1 |
| Cys | semisauer | 8,33 | 8,8–9,1 |
| Tyr | semisauer | 10,07 | 9,7–10,1 |
| Lys | basisch | 10,53 | 9,3–9,5 |
| Arg | basisch | 12,48 | – |
Aliphatische Aminosäureseitenketten
Aromatische Aminosäureseitenketten
Amidierte Aminosäureseitenketten
Schwefel-enthaltende Aminosäureseitenketten
Hydroxylierte Aminosäureseitenketten
Basische Aminosäureseitenketten
Saure Aminosäureseitenketten
- Asparaginsäure (in Wasser dissoziiert zu Aspartat)
- Glutaminsäure (in Wasser dissoziiert zu Glutamat)
Der pK-Wert ist der pH-Wert, bei dem die titrierbaren Gruppen zu gleichen Teilen protoniert und deprotoniert vorliegen. D. h., die titrierbare Gruppe liegt zu gleichen Teilen in ihrer basischen, wie in ihrer sauren Form vor (siehe auch: Henderson-Hasselbalch-Gleichung).
Es ist meist üblich, anstatt vom pKS vom pK zu sprechen, das heißt vom pK der Säure. In diesem Sinne müsste allerdings vom pK des Lysins als pKB, also vom pK der Base gesprochen werden. Aus Gründen der Vereinfachung wird diese Notation aber allgemein weggelassen, da sie sich auch aus dem Sinnzusammenhang selbst ergibt (d. h. ob die Gruppe als Base oder Säure wirkt).
Der pK ist keine Konstante, sondern hängt von der Temperatur, der Aktivität, der Ionenstärke und der unmittelbaren Umgebung der titrierbaren Gruppe ab und kann daher stark schwanken.
Ist der pH höher als der pK einer titrierbaren Gruppe, so liegt die titrierbare Gruppe in ihrer basischen (deprotonierten) Form vor. Ist der pH niedriger als der pK der titrierbaren Gruppe, so liegt die titrierbare Gruppe in ihrer sauren (protonierten) Form vor:
- Für Asp (pK = 3,86) bei pH 7: Die Seitenkette ist nahezu vollständig deprotoniert.
- Für Lys (pK= 10,53) bei pH 7: Die Seitenkette ist nahezu vollständig protoniert.
Die Seitenketten basischer Aminosäuren sind in ihrer protonierten (sauren) Form einfach positiv geladen und in ihrer deprotonierten (basischen) Form ungeladen. Die Seitenketten der sauren Aminosäuren (einschließlich Cystein und Tyrosin) sind in ihrer protonierten (sauren) Form ungeladen und in ihrer deprotonierten (basischen) Form einfach negativ geladen. Deswegen spielt der pH-Wert für die Eigenschaften der Seitenkette eine so wichtige Rolle, da das Verhalten der Seitenkette ein ganz anderes ist, wenn sie geladen bzw. ungeladen ist.
Die titrierbaren Seitenketten beeinflussen zum Beispiel das Löslichkeitsverhalten der entsprechenden Aminosäure. In polaren Lösungsmitteln gilt: Geladene Seitenketten machen die Aminosäure löslicher, ungeladene Seitenketten machen die Aminosäure unlöslicher.
In Proteinen kann das dazu führen, dass bestimmte Abschnitte hydrophiler oder hydrophober werden, wodurch die Faltung und damit die Aktivität von Enzymen vom pH-Wert abhängt. Durch stark saure oder basische Lösungen können Proteine deswegen auch denaturiert werden.
| Aminosäuren | pK2COOH | pK1COOH | Isoelektrischer Punkt |
pK1NH2 | pK2NH2 |
|---|---|---|---|---|---|
| Alanin | – | 2,3 | 6,1 | 9,9 | – |
| Arginin | – | 2,81 | 10,76 | 9,09 | 13,2 |
| Asparagin | – | 2,02 | 5,41 | 8,80 | – |
| Asparaginsäure | 3,65 | 1,88 | 2,85 | 9,60 | – |
| Cystein | 8,33* | 1,71 | 5,05 | 10,78 | – |
| Glutamin | – | 2,17 | 5,65 | 9,13 | – |
| Glutaminsäure | 4,25 | 2,19 | 3,22 | 9,67 | – |
| Glycin | – | 2,21 | 5,97 | 9,15 | – |
| Histidin | – | 1,78 | 7,47 | 8,97 | 5,97 |
| Isoleucin | – | 2,32 | 5,94 | 9,76 | – |
| Leucin | – | 2,4 | 5,98 | 9,6 | – |
| Lysin | – | 2,20 | 9,59 | 8,90 | 10,28 |
| Methionin | – | 2,28 | 5,74 | 9,21 | – |
| Phenylalanin | – | 2,58 | 5,84 | 9,24 | – |
| Prolin | – | 1,99 | 6,3 | 10,60 | – |
| Serin | – | 2,21 | 5,68 | 9,15 | – |
| Threonin | – | 2,10 | 5,60 | 9,12 | – |
| Tryptophan | – | 2,15 | 5,64 | 9,12 | – |
| Tyrosin | 10,07** | 2,20 | 5,66 | 9,11 | – |
| Valin | – | 2,30 | 5,96 | 9,60 | – |
| * Thiolgruppe ** phenolische Hydroxygruppe | |||||
Tabellenübersicht der Eigenschaften
| Aminosäure | Abk. | Code | Aminosäure-Seitenkette (R) | Charakter von R | Molare Masse von R (g/mol) | van-der-Waals-Volumen von R | Polarität von R | Hydro- phobi- zität[26] von R |
Acidität oder Basizität von R | Säure-Konstante (pKs) von R |
Struktur- formel |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Alanin | Ala | A | –CH3 | aliphatisch | 15,03 | 67 | unpolar | 1,8 | neutral | – | |
| Arginin | Arg | R | –CH2CH2CH2NH-C(NH)NH2 | aliphatisch | 100,14 | 148 | polar | −4,5 | basisch (stark) | 12,48 | |
| Asparagin | Asn | N | –CH2CONH2 | aliphatisch | 58,06 | 96 | polar | −3,5 | neutral | – | |
| Asparaginsäure | Asp | D | –CH2COOH | aliphatisch | 59,04 | 91 | polar | −3,5 | sauer | 3,90 | |
| Cystein | Cys | C | –CH2SH | aliphatisch | 47,10 | 86 | polar | 2,5 | neutral | 8,18 | |
| Glutamin | Gln | Q | –CH2CH2CONH2 | aliphatisch | 72,09 | 114 | polar | −3,5 | neutral | – | |
| Glutaminsäure | Glu | E | –CH2CH2COOH | aliphatisch | 73,07 | 109 | polar | −3,5 | sauer | 4,07 | |
| Glycin | Gly | G | –H | aliphatisch | 1,01 | 48 | unpolar | −0,4 | neutral | – | |
| Histidin | His | H | –CH2(C3H3N2) | aromatisch | 81,1 | 118 | polar | −3,2 | basisch (schwach) | 6,04 | |
| Isoleucin | Ile | I | –CH(CH3)CH2CH3 | aliphatisch | 57,11 | 124 | unpolar | 4,5 | neutral | – | |
| Leucin | Leu | L | –CH2CH(CH3)2 | aliphatisch | 57,11 | 124 | unpolar | 3,8 | neutral | – | |
| Lysin | Lys | K | –CH2CH2CH2CH2NH2 | aliphatisch | 72,13 | 135 | polar | −3,9 | basisch | 10,54 | |
| Methionin | Met | M | –CH2CH2SCH3 | aliphatisch | 75,15 | 124 | unpolar | 1,9 | neutral | – | |
| Phenylalanin | Phe | F | –CH2(C6H5) | aromatisch | 91,13 | 135 | unpolar | 2,8 | neutral | – | |
| Prolin | Pro | P | Anmerkung[27] | heterocyclisch | 42,08 | 90 | unpolar | −1,6 | neutral | – | |
| Serin | Ser | S | –CH2OH | aliphatisch | 31,03 | 73 | polar | −0,8 | neutral | – | |
| Threonin | Thr | T | –CH(OH)CH3 | aliphatisch | 45,06 | 93 | polar | −0,7 | neutral | – | |
| Tryptophan | Trp | W | –CH2(C8H6N) | aromatisch | 130,16 | 163 | unpolar | −0,9 | neutral | – | |
| Tyrosin | Tyr | Y | –CH2(C6H4)OH | aromatisch | 107,13 | 141 | polar | −1,3 | neutral | 10,46 | |
| Valin | Val | V | –CH(CH3)2 | aliphatisch | 43,09 | 105 | unpolar | 4,2 | neutral | – |
Stereochemie
18 der 20 proteinogenen Aminosäuren haben gemäß der Cahn-Ingold-Prelog-Konvention am α-Kohlenstoff-Atom die (S)-Konfiguration, lediglich Cystein besitzt die (R)-Konfiguration, da hier der Kohlenstoff mit der Thiolgruppe eine höhere Priorität als die Carbonsäuregruppe hat. Glycin ist nicht chiral, daher kann keine absolute Konfiguration bestimmt werden.
Zusätzlich zum Stereozentrum am α-C-Atom besitzen Isoleucin und Threonin in ihrem Rest R je ein weiteres stereogenes Zentrum. Proteinogenes Isoleucin [R = –C*H(CH3)CH2CH3] ist dort (S)-konfiguriert, Threonin [R = –C*H(OH)CH3] (R)-konfiguriert.
Nichtproteinogene Aminosäuren
Von den nichtproteinogenen, also den nicht während der Translation in Proteine eingebauten, Aminosäuren sind bislang über 400 bekannt, die in Organismen vorkommen.[2] Dazu gehört etwa das L-Thyroxin, ein Hormon der Schilddrüse, L-DOPA, L-Ornithin oder das in fast allen Arten von Cyanobakterien nachgewiesene Neurotoxin β-Methylaminoalanin (BMAA). Die L-Azetidin-2-carbonsäure ist ein toxischer Bestandteil der Rhizome einheimischer Maiglöckchen (Convallaria majalis) und Zuckerrüben. Sie wirkt hemmend auf das Pflanzenwachstum.
Die meisten nichtproteinogenen Aminosäuren leiten sich von den proteinogenen ab, die L-α-Aminosäuren sind. Dennoch können dabei auch β-Aminosäuren (β-Alanin) oder γ-Aminosäuren (GABA) entstehen.

Zu den nichtproteinogenen Aminosäuren zählen auch alle D-Enantiomere der proteinogenen L-Aminosäuren. D-Serin wird im Hirn durch die Serin-Racemase aus L-Serin (seinem Enantiomer) erzeugt. Es dient sowohl als Neurotransmitter als auch als Gliotransmitter durch die Aktivierung des NMDA-Rezeptors, was zusammen mit Glutamat die Öffnung des Kanals erlaubt. Zum Öffnen des Ionenkanals muss Glutamat und entweder Glycin oder D-Serin binden. D-Serin ist an der Glycin-Bindungsstelle des Glutamatrezeptors vom NMDA-Typ ein stärkerer Agonist als Glycin selbst, war aber zum Zeitpunkt der Erstbeschreibung der Glycin-Bindungsstelle noch unbekannt. D-Serin ist nach D-Aspartat die zweite D-Aminosäure, die in Menschen gefunden wurde.[28]
Zu den synthetischen Aminosäuren gehört die 2-Amino-5-phosphonovaleriansäure (APV), ein Antagonist des NMDA-Rezeptors und das ökonomisch wichtige D-Phenylglycin [Synonym: (R)-Phenylglycin], das in der Seitenkette vieler semisynthetischer β-Lactamantibiotica als Teilstruktur enthalten ist. (S)- und (R)-tert-Leucin [Synonym: (S)- und (R)-β-Methylvalin] sind synthetische Strukturisomere der proteinogenen Aminosäure (S)-Leucin und werden als Edukt in stereoselektiven Synthesen eingesetzt.
Es gibt auch Aminosulfonsäuren [Beispiel: 2-Aminoethansulfonsäure (Synonym: Taurin)], α-Aminophosphonsäuren und α-Aminophosphinsäuren.[29] Das sind auch α-Aminosäuren, jedoch keine α-Aminocarbonsäuren. Statt einer Carboxygruppe (–COOH) ist eine Sulfonsäure-, Phosphonsäure- bzw. Phosphinsäuregruppe in diesen α-Aminosäuren enthalten.
| Aminosäure | Biologische Bedeutung |
|---|---|
| Thyroxin | Hormon der Schilddrüse |
| GABA | inhibitorischer Neurotransmitter |
| L-Homoserin | Stoffwechselzwischenprodukt der Argininsynthese |
| Ornithin | Stoffwechselzwischenprodukt im Harnstoffzyklus |
| Citrullin | Stoffwechselzwischenprodukt im Harnstoffzyklus |
| Argininosuccinat | Stoffwechselzwischenprodukt im Harnstoffzyklus |
| L-DOPA | Stoffwechselzwischenprodukt bei der Synthese von Katecholaminen |
| 5-Hydroxytryptophan | Stoffwechselzwischenprodukt bei der Serotoninsynthese |
| β-Alanin | Baustein von Coenzym A |
| β-Methylamino-Alanin | Neurotoxin in Cyanobakterien |
| Ibotensäure | Pilzgift |
| D-Valin | Bestandteil des Antibiotikums Valinomycin |
| D-Alanin | Bestandteil in bakteriellen Zellwänden |
| D-Glutamat | Bestandteil in bakteriellen Zellwänden |
| 2,6-Diaminopimelinsäure | Bestandteil in bakteriellen Zellwänden |
Nachweis
Ein quantitativer photometrischer Nachweis von Aminosäuren kann unter anderem mit Ninhydrin oder dem Folin-Reagenz erfolgen.
Verwendung
Aminosäuren haben für die Ernährung des Menschen eine fundamentale Bedeutung. In der Regel wird der Bedarf an essentiellen Aminosäuren durch tierische oder eine geeignete Kombination verschiedener pflanzlicher Proteine (etwa aus Getreide und Hülsenfrüchten)[30] vollkommen gedeckt. Pflanzliche Proteine haben meist eine geringere biologische Wertigkeit. Futtermittel in der Nutztierhaltung werden zusätzlich mit Aminosäuren, z. B. DL-Methionin und L-Lysin, angereichert, wodurch deren Nährwert erhöht wird.
Aminosäuren bzw. ihre Derivate finden Verwendung als Zusatz für Lebensmittel, hier insbesondere auch als Geschmacksverstärker (Natriumglutamat), Süßstoff (Aspartam), und sind Vorstufen für bestimmte Aromastoffe, die bei der Zubereitung von Speisen durch die Maillard-Reaktion entstehen.
In der Pharmazie bzw. Medizin werden L-Aminosäure-Infusionslösungen für die parenterale Ernährung angewendet. Daneben werden Aminosäuren auch als Hilfsstoffe eingesetzt, z. B. als Salzbildner, Puffer und Stabilisatoren bei bestimmten Lebererkrankungen. Bei Krankheiten mit einem Mangel von Neurotransmittern verwendet man L-Dopa. Für synthetische Peptidhormone und für die Biosynthese von Antibiotika sind Aminosäuren notwendige Ausgangsstoffe. Magnesium- und Kalium-Aspartate spielen bei der Behandlung von Herz- und Kreislauferkrankungen eine Rolle. Cystein, beziehungsweise die Derivate Acetylcystein und Carbocystein, finden zudem eine Anwendung bei infektiösen Bronchialerkrankungen mit erhöhtem Bronchialsekret. Zudem wird L-Cystein als Reduktionsmittel in der Dauerwelle eingesetzt.[31]
Aminosäuren werden in der Kosmetik Hautpflegemitteln und Shampoos zugesetzt.
Siehe auch
- Aminosäure-Stoffwechsel
- Aminosäuredatierung, eine Methode zur Altersbestimmung fossiler Knochen
- Leucine
- Valine
Literatur
Bücher
- Harold Hart: Organische Chemie: Ein kurzes Lehrbuch. VCH, 1989, ISBN 3-527-26480-9.
- Lubert Stryer: Biochemie. 4. Auflage, Spektrum Akademischer Verlag, 1995, ISBN 3-86025-346-8.
- Roland Glaser: Biophysik. 4. Auflage, Gustav Fischer Verlag, 1996, ISBN 3-334-60967-7 (Fischer), ISBN 3-8252-8116-7.
- Uwe Meierhenrich: Amino Acids and the Asymmetry of Life. Springer-Verlag, Heidelberg und Berlin 2008, ISBN 978-3-540-76885-2.
- Friedrich Lottspeich, Haralabos Zorbas: Bioanalytik. Spektrum Akademischer Verlag, Heidelberg 1998, ISBN 978-3-8274-0041-3.
- Hubert Rehm, Thomas Letzel: Der Experimentator: Proteinbiochemie / Proteomics. 6. Auflage, Spektrum Akademischer Verlag, Heidelberg 2009, ISBN 978-3-8274-2312-2.
Zeitschriftenartikel
- Bing Hao, Weimin Gong, Tsuneo K. Ferguson, Carey M. James, Joseph A. Krzycki, Michael K. Chan: A New UAG-Encoded Residue in the structure of a Methanogen Methyltransferase. In: Science 296, 2002, S. 1462–1466.
- S. J. Freeland, L. D. Hurst: Der raffinierte Code des Lebens. In: Spektrum der Wissenschaft Nr. 6, 2004, S. 86–93.
- Lei Wang, Peter G. Schultz: Die Erweiterung des genetischen Codes. In: Angewandte Chemie 117, Nr. 1, 2005, S. 34–68.
- Wolfgang Leuchtenberger, Ulf Plöcker: Herstellung von Aminosäuren mit Hilfe biotechnologischer Methoden. In: Chemie-Ingenieur-Technik 60, 1988, S. 16–23.
- Bernd Hoppe, Jürgen Martens: Aminosäuren – Bausteine des Lebens, Chemie in unserer Zeit, 17. Jahrg. 1983, Nr. 2, S. 41–53.
- Bernd Hoppe, Jürgen Martens: Aminosäuren – Herstellung und Gewinnung, Chemie in unserer Zeit, 18. Jahrg. 1984, Nr. 3, S. 73–86.
Weblinks
Einzelnachweise
- ↑ a b , Katharina Munk (HRsg.): Biochemie – Zellbiologie. Georg Thieme Verlag, Stuttgart 2008, ISBN 3131519916, S. 126, Google Books
- ↑ a b Peter Nuhn: Naturstoffchemie, S. Hirzel Wissenschaftliche Verlagsgesellschaft Stuttgart, 1990, S. 70, ISBN 3-7776-0473-9.
- ↑ NASA Researchers Make First Discovery of Life’s Building Block in Comet. nasa.gov, August 2009; Chiral amino acids in meteorites strengthen evidence for extraterrestrial life. spie.org, September 2010 (abgerufen am 4. Oktober 2010).
- ↑ Leben im All: Teleskop Alma findet komplexe Moleküle im All. Abgerufen am 29. September 2014.
- ↑ L. Vauquelin, P. Robiquet: The discovery of a new plant principle in Asparagus sativus. In: Annales de Chimie. Band 57, 1806, S. 88–93.
- ↑ W. Rose et al.: Feeding Experiments with Mixtures of Highly Purified Amino Acids. VIII. Isolation and Identification of a New Essential Amino Acid. In: Journal of Biological Chemistry. Band 112, 1935, S. 283–302.
- ↑ R. Simoni, R. Hill, M. Vaughan: The Discovery of the Amino Acid Threonine: the Work of William C. Rose. In: Journal of Biological Chemistry. Band 277, Nr. 37, 13 September 2002, S. 56–58.
- ↑ S. Hansen: Die Entdeckung der proteinogenen Aminosäuren von 1805 in Paris bis 1935 in Illinois. Berlin 2015.
- ↑ a b c L. F. Fieser, M. Fieser: Lehrbuch der organischen Chemie. 3. Auflage, Verlag Chemie, 1957, S. 506.
- ↑ a b L. F. Fieser, M. Fieser: Lehrbuch der organischen Chemie. 3. Auflage, Verlag Chemie, 1957, S. 507.
- ↑ L. F. Fieser, M. Fieser: Lehrbuch der organischen Chemie. 3. Auflage, Verlag Chemie, 1957, S. 511.
- ↑ L. F. Fieser, M. Fieser: Lehrbuch der organischen Chemie. 3. Auflage, Verlag Chemie, 1957, S. 516.
- ↑ a b L. F. Fieser, M. Fieser: Lehrbuch der organischen Chemie. 3. Auflage, Verlag Chemie, 1957, S. 508.
- ↑ a b L. F. Fieser, M. Fieser: Lehrbuch der organischen Chemie. 3. Auflage, Verlag Chemie, 1957, S. 510.
- ↑ Bernd Hoppe, Jürgen Martens: Aminosäuren – Herstellung und Gewinnung, Chemie in unserer Zeit, 18. Jahrg. 1984, Nr. 3, S. 73–86.
- ↑ Siegfried Hauptmann: Organische Chemie, 2. durchgesehene Auflage, VEB Deutscher Verlag für Grundstoffindustrie, Leipzig, 1985, S. 506–507, ISBN 3-342-00280-8.
- ↑ a b Wissenschaft-Online-Lexika: Eintrag zu Aminosäuren im Lexikon der Biologie, abgerufen am 25. April 2009.
- ↑ G. Löffler, P. E. Petrides und P. C. Heinrich: Biochemie & Pathobiochemie. 8. Auflage, Springer, Heidelberg 2006, ISBN 978-3-540-32680-9.
- ↑ a b International Union of Pure and Applied Chemistry and International Union of Biochemistry: Nomenclature and Symbolism for Amino Acids and Peptides (Recommendations 1983). In: Pure & Appl. Chem. Band 56, Nr. 5, 1984, S. 595–624, doi:10.1351/pac198456050595.
- ↑ Paula Yurkanis Bruice: Organic Chemistry. Pearson Education Inc., 2004, 4. Auflage, S. 960–962, ISBN 0-13-121730-5.
- ↑ Katsura Asano: Why is start codon selection so precise in eukaryotes?. In: Translation. Band 2, Nr. 1, März 2014, doi:10.4161/trla.28387, PMC 4705826 (freier Volltext).
- ↑ Kathrin Lang, Lloyd Davis u. a.: Genetically encoded norbornene directs site-specific cellular protein labelling via a rapid bioorthogonal reaction. In: Nature Chemistry. 2012, S. 298–304, doi:10.1038/nchem.1250.
- ↑ a b c W. R. Taylor: The classification of amino acid conservation. In: Journal of Theoretical Biology. Band 119, Jahrgang 1986, S. 205–218. doi:10.1016/S0022-5193(86)80075-3.
- ↑ Löffler: Basiswissen Biochemie. Springer-Lehrbuch, Heidelberg 2005, ISBN 3-540-23885-9, S. 24.
- ↑ Alberts, Johnson, Lewis, Raff, Roberts, Walter: Molekularbiologie der Zelle. WILEY-VCH Verlag GmbH, Weinheim 2004, ISBN 3-527-30492-4, S. 152.
- ↑ J. Kyte, R. F. Doolittle: A simple method for displaying the hydropathic character of a protein. In: Journal of Molecular Biology. Band 157, Nr. 1, 1982, S. 105–132, PMID 7108955.
- ↑ Darstellung nicht verfügbar, da bei Prolin am Peptid-Rückgrat ein H-Atom am Stickstoff weniger vorkommt (ein sekundäres Amin), weil die Seitenkette mit dem Stickstoffatom einen Ring bildet (–NHCH2CH2CH2–).
- ↑ Jean-Pierre Mothet, Angèle T. Parent, Herman Wolosker, Roscoe O., Jr. Brady, David J. Linden, Christopher D. Ferris, Michael A. Rogawski, Solomon H. Snyder: d-Serine is an endogenous ligand for the glycine site of the N-methyl-d-aspartate receptor. In: Proc. Natl. Acad. Sci. USA. 97. Jahrgang, Nr. 9, 2000, S. 4926–4931, doi:10.1073/pnas.97.9.4926, PMID 10781100, PMC 18334 (freier Volltext).
- ↑ Karlheinz Drauz, Hans Günter Koban, Jürgen Martens, Werner Schwarze: Phosphonic and Phosphinic Acid Analogs of Penicillamine. In: Liebigs Annalen der Chemie. Band 1985, Nr. 3, 1985, S. 448–452, doi:10.1002/jlac.198519850303.
- ↑ Eintrag zu Proteinwertigkeit. In: Römpp Online. Georg Thieme Verlag
- ↑ Wolfgang Legrum: Riechstoffe, Zwischen Gestank Und Duft: Vorkommen, Eigenschaften und Anwendung von Riechstoffen und deren Gemischen, Gabler Wissenschaftsverlage (2011), S. 165 (eingeschränkte Vorschau in der Google-Buchsuche).






















